Des scientifiques de l’Institut Pasteur à Paris, du CNRS, de l’Inserm, de l’Institut Pasteur de Lille, de l’Université Lille 2, en collaboration avec le CEA-Genoscope et le Sanger Institute, viennent de déterminer l’origine de l’émergence de la bactérie Mycobacterium tuberculosis, principal agent de la tuberculose. Les chercheurs apportent également des indices sur les raisons de son succès évolutif. Ils ont identifié plusieurs mécanismes génétiques ayant pu contribuer à la dissémination mondiale du pathogène, qui infecte actuellement jusqu’à 2 milliards d’individus. Ces travaux, publiés online le 6 janvier sur le site de Nature Genetics, ouvrent des perspectives pour identifier de nouvelles cibles pour lutter contre la tuberculose.
Communiqué de presse
Paris, le 7 janvier 2013
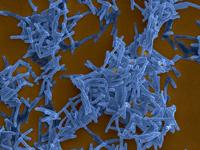
Les équipes de Roland Brosch , à l’Institut Pasteur à Paris et de Philip Supply , à l’Institut Pasteur de Lille, en collaboration avec le CEA-Genoscope et le Sanger Institute, sont parvenues à retracer l’histoire évolutive de M. tuberculosis. Leur étude a permis de confirmer l'hypothèse de précédents travaux suggérant que les souches de M. tuberculosis, génétiquement très conservées, sont issues d'une branche évolutive commune aux souches de Mycobacterium canettii (1) (M. canettii). Ces dernières, qui entraînent également la tuberculose, présentent une très large diversité génétique et certaines caractéristiques de leurs génomes indiquent qu'elles sont d’une origine beaucoup plus ancienne. Les scientifiques fournissent également des indications possibles sur les facteurs qui ont contribué au succès évolutif de M. tuberculosis, qui a fait de la tuberculose une pandémie mondiale, alors que les souches de M. canettii sont restées majoritairement cantonnées dans les régions de l'Est de l'Afrique.
L’équipe de chercheurs, menée par Philip Supply et Roland Brosch, suggère notamment que les souches de M. tuberculosis ont acquis leur virulence et leur persistance par une combinaison de plusieurs mécanismes génétiques, tels que : une perte de fonction de certains gènes ou encore l’acquisition de nouveaux gènes par transfert horizontal. Grâce à l’identification de gènes possiblement impliqués chez M. tuberculosis, l’ensemble de ces travaux ouvre des perspectives pour la lutte contre la tuberculose, dont l’OMS a fait l’une de ses priorités.
(1) Décrit pour la première fois en 1969 par le microbiologiste français Georges Canetti, chercheur à l’Institut Pasteur qui consacra sa vie à l’étude de la tuberculose. Chaque année, le prix Georges, Jacques et Elias Canetti récompense des chercheurs de l'Institut Pasteur.
--
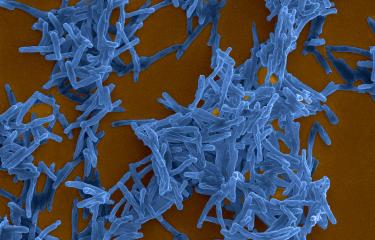
Illustration - Copyright Institut Pasteur
Légende - Mycobacterium tuberculosis, agent de la tuberculose. Microscopie électronique à balayage
Sources
Genome analysis of smooth tubercle bacilli provides insights into ancestry and pathoadaptation of Mycobacterium tuberculosis, Nature Genetics, january 6, 2013.
Philip Supply (1,2,3,4), Michael Marceau (1,2,3,4), Sophie Mangenot (5), David Roche (5,6), Carine Rouanet (1,2,3,4), Varun Khanna (7), Laleh Majlessi (8,9), Alexis Criscuolo (10), Julien Tap (10), Alexandre Pawlik (7), Laurence Fiette (11,12), Mickael Orgeur (7), Michel Fabre (13), Cécile Parmentier (7), Wafa Frigui (7), Roxane Simeone (7), Eva C. Boritsch (7), Anne-Sophie Debrie (1,2,3,4), Eve Willery (1,2,3,4), Danielle Walker (14), Michael A. Quail (14), Laurence Ma (15), Christiane Bouchier (15), Grégory Salvignol (5,6), Fadel Sayes (8,9), Alessandro Cascioferro (7), Torsten Seemann (16), Valérie Barbe (5), Camille Locht (1,2,3,4), Maria-Cristina Gutierrez (1,2,3,4,17), Claude Leclerc (8,9), Stephen D. Bentley (14), Timothy P. Stinear (18), Sylvain Brisse (10), Claudine Médigue (5,6), Julian Parkhill (14), Stéphane Cruveiller (5,6) & Roland Brosch (7).
(1) Institut National de la Santé et de la Recherche Médicale (INSERM), U1019, Center for Infection and Immunity of Lille, Lille, France;
(2) Centre National de la Recherche Scientifique (CNRS), Unite mixte de recherche (UMR) 8204, Center for Infection and Immunity of Lille, Lille, France;
(3) Univ Lille Nord de France, Université Lille 2, Center for Infection and Immunity of Lille, Lille, France;
(4) Institut Pasteur de Lille, Center for Infection and Immunity of Lille, Lille, France;
(5) Commissariat à l'Energie Atomique et aux Energies Alternatives CEA/DSV/IG/Genoscope, LABGeM, Evry, France;
(6) CNRS-UMR 8030 , Evry, France;
(7) Institut Pasteur, Unit for Integrated Mycobacterial Pathogenomics, Paris, France;
(8) Institut Pasteur, Unité de Régulation Immunitaire et Vaccinologie, Paris, France;
(9) INSERM U1041, Paris, France;
(10) Institut Pasteur, Genotyping of Pathogens and Public Health (PF8), Paris, France;
(11) Institut Pasteur, Unité d'Histopathologie Humaine et Modèles Animaux, Paris, France;
(12) Université Versailles-Saint Quentin en Yvelines, Faculté de Médecine, DER Histologie, Versailles, France;
(13) Laboratoire de Biologie Clinique, HIA Percy, Clamart, France;
(14) Wellcome Trust Sanger Institute, Hinxton, Cambridge, UK;
(15) Institut Pasteur, Genopole, Platform Genomics PF1, Paris, France;
(16) Victorian Bioinformatics Consortium, Monash University, Clayton, Australia;
(17) Institut Pasteur, Department d’Infection et d’Epidemiologie, Paris, France;
(18) Department of Microbiology and Immunology, University of Melbourne, Parkville, Australia.
Contact presse
Service de presse de l'Institut Pasteur
Nadine Peyrolo - + 33 (0)1 45 68 81 46
Jérémy Lescène - +33 (0)1 45 68 81 01
presse@pasteur.fr
Institut Pasteur Lille service de communication
Marie-José Hermant - + 33 (0)3 20 87 78 08
marie-jose.hermant@pasteur-lille.fr
Eugénie Devendeville - + 33 (0)3 20 87 77 38
eugenie.dumeignil@pasteur-lille.fr