- EN
- FR
- EN
- FR

Activités du CNR Listeria
Avant tout envoi, consulter la rubrique Envoyer un échantillon/une souche
Toute demande au CNR n’est prise en compte que si elle est accompagnée des renseignements, notamment cliniques, à mentionner sur la feuille de demande d'analyse fournie par le CNR et devant accompagner l’échantillon.
Fiche de renseignements pour les souches d’origine humaine (PDF - 199 Ko)
Fiche de renseignement pour les souches d’origine non humaine (PDF - 213 Ko)
Le CNR Listeria réalise ses activités analytiques sous management de la qualité selon la norme NF EN ISO 15189 version 2012.
Le CNR poursuit la démarche d’accréditation selon les conditions imposées par la Loi du 30 mai 2013 (2013-442).
Déclaration Commission Nationale sur l’Informatique et les Libertés (Loi CNIL n°78-17 du 06 janvier 1978) N° 1474696 v0
ANALYSES
Le CNRL effectue également des analyses d'identification et de typage moléculaire dans le cadre de prestations privées en agro-alimentaire.
Le CNRL n’effectue pas d’analyses de 1ère intention (en conformité avec l’arrêté du 16 Juin 2016, sauf à la demande de l’ARS, de SPF ou de la DGS dans le cadre d’expertises) mais des analyses de confirmation pour la confirmation du cas de listériose et/ou de l’alerte produit.
Le CNRL ne réalise pas (i) de sérologie, compte tenu de l’intérêt en pratique clinique non démontré de cette technique et des recommandations du REMIC, ou (ii) de PCR ou qPCR sur LCR ou d’autres échantillons cliniques à visée diagnostic, qui sont effectuées en LABM de 1er intention.
La PCR sur tout échantillon biologique (LCR, liquide amniotique, etc.) est communiquée au service de microbiologie de l’Hôpital Necker Enfants Malades en accord avec Santé Publique France (SPF).
Après le contrôle à réception de la conformité de l’envoi au contrat de prestation et à la vérification de la pureté :
1) Identification du genre et de l’espèce par spectrométrie de masse Maldi-Tof (Bruker Daltonics, Allemagne) et recherche du caractère hémolytique, complétés par d’autres tests classiques si nécessaire (Mode opératoire SOP RL 00040 version informatique en vigueur). La technique de spectrométrie de masse Maldi-Tof a été validée par l’analyse de plus de 517 souches identifiées préalablement par la méthode traditionnelle biochimique et le séquençage génomique.
Les tests biochimiques [galerie API-Listeria® (bioMérieux, France)] ne sont utilisés qu’exceptionnellement, en cas de panne du spectromètre de masse de l’obtention de résultats ambigus ou de nécessité de détermination de la sous-espèce. En cas de besoin, les souches atypiques ou identifiées comme non Listeria spp. sont identifiées par analyse de la séquence du gène codant pour la sous-unité ribosomale 16S ou du gène iap.
2) Détermination du sérogroupe PCR (Mode opératoire SOP RL 000135 version informatique en vigueur) selon la méthode publiée par le CNRL en 2004(Doumith et al., 2004) et amendée en 2011 (Leclercq et al., 2011). Cette PCR multiplex cible une partie de la séquence du gène prs présent dans l’ensemble des espèces décrites de Listeria et quatre autres gènes (Lmo1118, Lmo0737, ORF2110, ORF2819) spécifiques de Lm, permettant de déterminer le sérogroupe PCR.
3) Antibiogramme de toutes les souches d’origine humaine en utilisant la technique de dilution en milieu gélosé selon les recommandations de l’EUCAST (Mode opératoire SOP RL 000142 version informatique en vigueur). Ces antibiogrammes sont effectués sur un panel de 23 antibiotiques. La lecture de l’antibiogramme est réalisée sur un automate paramétré pour le référentiel EUCAST. Les éventuelles résistances sont confirmées par la détermination de la CMI par E-test. Les mécanismes des résistances identifiées sont ensuite étudiés en collaboration avec le CNR de la Résistance aux Antibiotiques.
4) Séquençage du génome (Mode opératoire SOP RL 000198 version informatique en vigueur). Les ADNs génomiques sont extraits (méthode DNeasy Blood & Tissue extraction kit (Qiagen, Danemark)), et leur qualité est vérifiée par fluorimétrie. La préparation des librairies est réalisée en utilisant le kit NEXTERA XT DNA Sample et les séquences génomiques sont déterminées sur la plateforme Illumina NextSeq 500 (Illumina, Californie, USA). L’assemblage est réalisé avec le logiciel CLC Assembly Cell 4.3.0 (Qiagen, Danemark). Le cgMLST (Moura et al., 2016, 2017) est déduit du génome assemblé par l’algorithme BLASTN implémenté dans la plateforme BIGSdb-Lm. Les profils cgMLST obtenus sont comparés et analysés dans le logiciel bioNumerics version 7.6.2. Le cgMLST est utilisé en routine depuis le 1er janvier 2017 pour la surveillance, en remplacement de la PFGE. L’analyse des génomes permet également la détection de gènes de virulence et de résistance aux antibiotiques et antiseptiques. De plus, les complexes clonaux MLST (Ragon et al., 2008) auxquels appartiennent les souches peuvent être déduits des séquences génomiques, permettant d’estimer le niveau de virulence de ces souches (Maury, Tsai et al., 2016).
EXPERTISES
Des expertises complémentaires peuvent également être effectuées, dans le cadre de projets de recherche ou pour approfondir une investigation dans le cadre de la surveillance nationale:
- Typage MLST 7 gènes (Ragon et al., 2008). Ce typage permet de comparer rapidement les souches par rapport aux complexes clonaux qui ont déjà été à l’origine d’épidémies ou de cas groupés et de prédire leur potentiel infectieux (Maury, Tsai et al., 2016). Notre base de données MLST est disponible à http://bigsdb.pasteur.fr/listeria/.
- Typage MLST par PCR multiplexe. L’appartenance à un clone MLST peut également être déterminée rapidement et avec une bonne fiabilité par une méthode de PCR multiplexe dite de « clonogrouping » (Chenal-Francisque, Maury et al., 2015). Celle-ci a été développée et brevetée par le CNRL.
- Caractérisation de la virulence des souches de Lm par inoculation par voie orale ou parentérale de gerbilles ou de souris humanisées et/ou par des tests in vitro.
- A la demande de l’ANSM ou de SPF, le CNRL réalise également des analyses de détection de souches de Lm viables par isolement sur gélose ALOA (bioMérieux) à partir d’échantillons de selles.
Des expertises peuvent également être effectuées sur des prélèvements biologiques comme des selles pour détecter les Listeria spp. ou Listeria monocytogenes. Dans ce dernier cas, veuillez contacter le CNR à listeria@pasteur.fr pour l’envoi des échantillons de produits biologiques nécessitants le consentement du patient pour les analyses réalisées.
SURVEILLANCE
Surveillance de la listériose humaine en France
Le CNRL est en charge de la surveillance microbiologique de la listériose et participe aux investigations destinées à identifier l’origine alimentaire des cas (Figure 1). Cette surveillance s’effectue en lien avec Santé Publique France (SPF), la Direction Générale de la Santé (DGS), la Direction Générale de l’Alimentation (DGAl) et la Direction Générale de la Consommation, de la Concurrence et de la Répression des Fraudes (DGCCRF), qui composent depuis 1992 la cellule Listeria. Depuis 2007, cette cellule comprend également le Laboratoire National de Référence des Listeria monocytogenes (LNRl) situé au laboratoire de Santé animale de l’Agence nationale de sécurité sanitaire de l’alimentation, de l’environnement et du travail (ANSES-LSA).
Les missions de la cellule sont (i) la détection des cas groupés de listériose, (ii) la proposition et la coordination des investigations et actions à mettre en œuvre devant des cas groupés potentiellement liés à une source commune de contamination, (iii) leur gestion et leur prévention.
Figure 1. Schéma de fonctionnement de la cellule Listeria
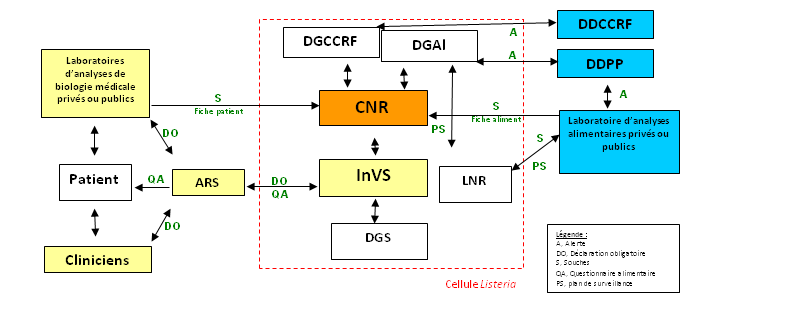
CNR : Centre national de Référence Listeria ; LNR : Laboratoire National de référence ; SPF : Santé Publique Franc; DGAl : Direction Générale de l’Alimentation ; DDPP : Directions Départementales de la Protection des Populations ; DDCCRF : Direction Départementales de la Consommation et de la Répression des Fraudes ; DGCCRF : Direction Générale de la Consommation et de la Répression des Fraudes ; ARS : Agence Régionale de Santé ; DGS : Direction Générale de la Santé.
Au sein de la « Cellule Listeria », le CNRL joue un rôle scientifique, technique et d’aide à la décision. Le circuit des souches d’origine alimentaire ou environnementale a été formalisé par la cellule Listeria comme présenté dans la Figure 2.
Figure 2. Circuit des souches sur le schéma de surveillance microbiologique français de L. monocytogenes (Source : BEH. 2012. Surveillance des L. monocytogenes dans les aliments. Hors Série : 41-45)
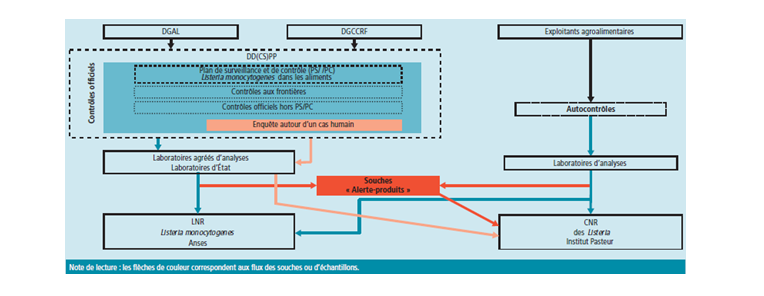
Surveillance microbiologique de la listériose en France
Le diagnostic de listériose repose sur l’isolement de Lm à partir d’un prélèvement biologique physiologiquement stérile ou de prélèvements périnataux.
La surveillance effectuée par le CNRL se fonde sur la caractérisation des souches isolées de patients et adressées volontairement par les biologistes, en parallèle à la déclaration obligatoire envoyée à l’ARS. Les cas diagnostiqués par une autre technique (biologie moléculaire ou sérologie) ne sont à ce jour pas retenus dans le cadre de la DO, car ces méthodes n'ont pas été formellement validées. La PCR hly est cependant très spécifique et peut constituer une aide diagnostique pour le clinicien, notamment dans les formes neuroméningées. Ce n’est pas le cas des tests sérologiques qui ne sont pas recommandés par le référentiel en Microbiologie (REMIC) en vigueur. Le CNRL caractérise également les souches alimentaires et environnementales qui lui sont adressées dans le cadre du système de surveillance.
Surveillance et dépassement de seuil
Plusieurs étapes successives d'alerte étaient définies jusqu’à décembre 2016. Ce schéma d’alerte a évolué en Janvier 2017 sur la base de l’analyse génomique cgMLST, qui remplace la macrorestriction d’ADN (PFGE) :
1. Définition de cluster dans le cadre de l’analyse génomique
Un cluster de souches est défini par la mise en évidence d’au moins deux souches, dont au moins une souche humaine depuis 2015, ayant une similarité de plus de 99,60% (7 allèles différents maximum sur les 1748 gènes du schéma cgMLST).
2. Surveillance microbiologique hebdomadaire
Le CNRL effectue chaque semaine un bilan des clusters cgMLST, qu’il communique par courrier électronique aux autres membres de la Cellule Listeria.
3. Surveillance renforcée
Tout cluster génomique est suivi d’une phase de surveillance renforcée durant laquelle le CNRL signale toute nouvelle souche d’origine humaine ou non humaine dont le profil cgMLST est similaire à celui du cluster génomique en question. SPF conduit les questionnaires alimentaires et les enquêtes épidémiologiques appropriées. La Cellule Listeria décide, en fonction des résultats de ces investigations, des actions à mettre en œuvre.
4. La phase d’Alerte
La Cellule Listeria peut décider du passage en phase d'alerte, définie comme toute situation présentant une menace potentielle pour la santé publique et nécessitant la mise en œuvre d’investigations ou d’actions complémentaires, soit en raison du nombre de cas détectés, soit en raison des hypothèses sur l'origine de la contamination.
La Cellule Listeria propose les investigations complémentaires et les actions à mener. Toutes les souches de Lm isolées dans le cadre de ces investigations sont envoyées au CNRL. La phase d'Alerte est levée par la Cellule Listeria. Certaines mesures spécifiques de surveillance peuvent être maintenues après la levée de l'Alerte.
5. Cas sporadiques, groupés et épidémies
Une épidémie correspond à un excès de cas de listériose, liés à une souche définie, le plus souvent en rapport avec une exposition précise, identifiée ou non. La déclaration d’épidémie est décidée par la Cellule Listeria, et tient compte de la distribution des cas dans le temps et sur le territoire. De façon schématique, si les cas possiblement liés à une même source sont regroupés dans le temps et l’espace, ils sont considérés comme des cas groupés, alors que s’il existe une distribution relativement large dans le temps et l’espace, une épidémie pourra alors être déclarée. Afin d’harmoniser avec la définition anglo-saxonne « Outbreak », il a été convenu qu’une épidémie nécessite l’identification de la source alimentaire. Un cas isolé, survenant en dehors de cas groupés ou d’épidémie, est qualifié de sporadique. La très grande majorité des cas de listériose identifiés par la déclaration obligatoire en France sont sporadiques.
Surveillance alimentaire
Les alertes-produits
Une alerte produit est lancée quand une denrée alimentaire non-conforme aux critères microbiologiques de sécurité du règlement européen 2073/2005 modifié (100 UFC/g ou absence/présence) a été mise sur le marché et que des mesures de retrait et/ou rappel auprès du consommateur doivent être prises. Les alertes-produits peuvent être mises en place suite à des contrôles officiels, des autocontrôles effectués par les professionnels ou des plans de surveillance et de contrôle. En France, ces alertes-produits sont déclenchées en cas de présence de Lm au sein de tout produit mis sur le marché, à consommer en l’état et permettant la croissance de Listeria, quel que soit le résultat du dénombrement. Cette limite est plus stricte que celle prévue par le règlement européen 2073/2005 modifié (100 UFC/g). Le CNRL effectue chaque semaine un rapport transmis par courrier électronique aux membres de la Cellule Listeria, sous la forme d’un tableau de suivi des clusters cgMLST.
Les enquêtes autour d’un cas de forme neuroméningée
Depuis 2001, pour tous les cas d’infection neuroméningée (cas avec signes cliniques d’atteinte neuroméningée et souche de Lm isolée du sang ou du LCR), des prélèvements effectués à partir des aliments du réfrigérateur du patient ou de son environnement (avec l’accord du patient ou le cas échéant de sa famille) sont réalisés par la DDPP. Des prélèvements dans les lieux d’achats du patient par la DDPP ou la DDCCRF peuvent également être effectués. Le CNRL compare le profil cgMLST des souches isolées des aliments à celle(s) isolée(s) chez le patient afin d’identifier l’aliment à l’origine du cas.
Surveillance de la résistance aux antibiotiques
La surveillance de la résistance de Lm aux antibiotiques est effectuée pour 23 antibiotiques (céfotaxime, sulfonamides, kanamycine, clindamycine, rifampicine, tétracyline, erythromycine, chloramphénicol, ciprofloxacine, moxifloxacine, levofloxacine, linézolide, triméthroprime, gentamicine, acide fusidique, vancomycine, streptomycine, pénicilline g, ampicilline, amoxicilline, acide nalidixique, imipénème, fosfomycine), à la recherche de résistances naturelles ou acquises.
Détection et analyse des infections nosocomiales
Le caractère nosocomial des infections à Lm est difficile à établir, car la période d’incubation de la listériose peut être longue et la source alimentaire de l'infection est difficile à identifier.
Ce mode de contamination a été décrit :
- soit par des aliments consommés à l'hôpital,
- soit par transmission croisée, notamment par le biais de matériel contaminé par un enfant infecté (thermomètres, couveuses, huile de soins).
Le CNRL détecte par le biais d’un système d’alerte automatisée au sein de sa base de données les suspicions d'infections nosocomiales, définies par l’identification de plus de 2 cas en moins de 15 jours dans un même établissement de santé. Cette identification déclenche une notification à Santé Publique France et la comparaison des caractéristiques microbiologiques des souches concernées en termes de type cgMLST. Si les caractéristiques microbiologiques des souches sont identiques ou similaires, une enquête impliquant SPF, l’ARS, la DGAL/DDPP et le CNRL ainsi que le CLIN et l’hygiéniste hospitalier concernés est conduite pour identifier l’origine de l’infection.
Les cas de bactériémie ou de formes neuroméningées contractées chez des patients hospitalisés depuis plus de 15 jours et sans apport de nourriture extérieur entraîne une inspection de la cuisine hospitalière, et la réalisation de prélèvements alimentaires et de surface.
Surveillance microbiologique de la listériose en Europe
L’ECDC a mis en place un système volontaire d’investigations de cas groupés ou d’épidémies européens au moyen de la plateforme d’échanges EPIS (Epidemic Intelligence Information System), ouverte aux pays européens, mais également aux USA, au Canada, à l’Australie, la Nouvelle-Zélande, l’Afrique du Sud, la Turquie, l’Islande, la Norvège, le Liechtenstein et le Japon. Ce système permet un échange de données de typages et d’informations concernant les cas groupés et les épidémies.
La base européenne ECDC TESSY (European Surveillance System) regroupe les données épidémiologiques transférées de façon volontaire et non exhaustive par les agences de santé publique nationales. Cette base est complétée par TESSY MOL, dont l’objectif est de lier les données épidémiologiques des cas humains aux données microbiologiques correspondantes (génosérotypage et typage moléculaire PFGE) dans le but de rendre possible une surveillance microbiologique européenne et une détection précoce des cas groupés ou épidémies au sein des pays participants, en lien avec la plateforme EPIS.
Le CNRL participe activement au groupe de travail Listeria du réseau européen des maladies d’origine hydrique et alimentaire (FWD) et au « Workshop on whole-genome sequencing for food and waterborne pathogens » de l’ECDC.
Il contribue, par le biais de SPF, à la notification de données françaises dans la base ECDC TESSY (European Surveillance System), et participe ainsi à la surveillance des maladies infectieuses au niveau européen. Il participe également, en relation avec SPF, aux investigations de cas groupés ou épidémies européennes, nommées « Urgent Inquiries », et signalées via la base ECDC EPIS (Epidemic Intelligence Information System).
Il participe activement à l’exercice européen EFSA-ECDC, au projet ELITE (European Listeria Typing Excercise) et à la Base Line Study EFSA (étude sur la contamination des aliments prêts-à-consommer et les cas humains survenus sur la période 2010-début 2011).
Le Laboratoire de Référence de l’Union Européenne (LRUE) pour Lm coordonne un réseau de laboratoires nationaux de référence dont les méthodes de détection et d’énumération des Lm dans les échantillons de la chaine alimentaire et de l’environnement agro-alimentaire, et les méthodes de typage moléculaire PFGE ont été harmonisées. Une base de données, sous l’égide de la DG-SANCO de l’Union Européenne et de l'EFSA, a été créée.
Surveillance microbiologique internationale de la listériose
L’Unité de Biologie des Infections héberge le Centre Collaborateur de l’OMS des Listeria.
Dans le cadre de son mandat renouvelé en Novembre 2015 (Mandat 2015-2019), le CCOMS participe à la surveillance internationale de la listériose, à la collecte de données pour l’analyse des risques réalisée par l’OMS, à la surveillance de la résistance aux agents antimicrobiens des Listeria, et à la formation des personnels qui le contactent.
Le CCOMS peut constituer un appui scientifique et technique pour l’OMS et la FAO en cas d’épidémies ou de cas groupés au niveau international, et émettre des recommandations, avis ou rédiger des documents d’informations.
COLLECTION DE SOUCHES
Les collections de souches du CNRL/CCOMS sont les suivantes :
Collection de souches types des espèces de Listeria et souches de référence pour la sérotypieLe CNRL dispose des souches types complètement caractérisées des 20 espèces et 6 sous-espèces du genre Listeria (L. monocytogenes, L. ivanovii subsp. ivanovii, L. ivanovii subsp. londoniensis, L. innocua, L. seeligeri, L. welshimeri, L. grayi subsp. grayi, L. grayi subsp. murrayi, L. rocourtiae, L. marthii, L. weihenstephanensis, L. fleischmannii subsp. fleischmannii, L. fleischmannii subsp. colorendiensis, Listeria floridensis, Listeria aquatica, Listeria cornellensis, Listeria riparia, Listeria grandensis, Listeria booriae, Listeria newyorkensis, L. goaensis, L. costaricensis, L. thailandensis), ainsi que des 15 souches de référence pour la sérotypie.
Collection de Listeria de l’Institut Pasteur (CLIP)
Chaque année, la collection du CNRL et du CCOMS s’incrémente d’environ 2000 souches caractérisées par phénotypie, génosérotypage, et génomique. Une base de données Lagon regroupe l’ensemble des métadonnées sur ces souches.
Cette collection majoritairement française, mais également internationale (collecte de souches dans le cadre du CCOMS), comportait environ 107.684 souches à la fin de l’année 2016. Ces souches sont d’origine clinique, alimentaire et environnementale, ainsi que vétérinaire ou de recherche.
Special Listeria Culture Collection (SLCC)
Il s’agit de la collection de Listeria d’H.P.R. Seeliger (Würzburg, Allemagne) qui comporte plus de 5.000 souches isolées entre 1926 et 1985. L’intérêt de cette collection léguée au CCOMS Listeria est qu’elle contient des souches anciennes, isolées depuis la première souche de L. monocytogenes identifiée (1921), de diverses origines géographiques (majoritairement en France et en Allemagne). Une base de données regroupe l’ensemble des informations disponibles sur ces souches.
Collection du Centre de Ressource Biologique de l’Institut Pasteur (CRBIP) comprenant la Collection de l’Institut Pasteur
Il s’agit d’une collection sous management de la qualité (certifiée AFAQ NF 96 900) dans laquelle le CNRL a déposé sous contrat 152 souches depuis 2004, qui constituent un échantillon représentatif des souches françaises humaines et non humaines du genre Listeria de 1981 à nos jours (souches d’épidémies, de travaux de séquençage, de sérovars rares, de référence, types, etc.). Des souches du genre Listeria et de l’espèce L. monocytogenes ayant des propriétés atypiques sont régulièrement mises en collection. La liste de ces souches est consultable à l’adresse web : https://catalogue-crbip.pasteur.fr/crbip_catalogue/faces/recherche_catalogue.xhtml et elles sont disponibles moyennant une somme couvrant les frais d’envoi et de maintien en collection sous assurance qualité.
Collection de sérums antifacteurs
Le CNRL produisait les 13 sérums contre les antigènes somatiques de Listeria, et si nécessaire les 5 sérums anti-flagellaires, utilisés pour la caractérisation antigénique des souches de L. monocytogenes et des autres espèces de Listeria.
Collection de bactériophages
Le CNRL possède la collection de bactériophages de lysotypie du Centre International de Lystopie des Listeria (1982-1992 ; Institut Pasteur, Paris).
Les souches type ainsi que les souches de référence pour la sérotypie peuvent être obtenues sur simple demande ou auprès de la C.I.P. (Collection de l’Institut Pasteur).
Toute commande de souches par le client auprès du CNR des Listeria donne lieu à la signature d’un accord de transfert de matériel biologique.
FORMATION /STAGE
Le CNRL peut participer, après étude des demandes, à des formations professionnelles ou des conférences/séminaires sur la listériose ou les Listeria.
Le CNRL peut accueillir après étude des demandes des stagiaires d’une période de moins de 3 mois (Stage de type M2, qualité, bioinformatique, etc.)
Veuillez adresser vos demandes à : listeria@pasteur.fr
COLLABORATIONS ET RÉSEAUX
Au niveau national
- SPF (Santé Publique France),
- la DGS (Direction générale à la santé),
- la DGAL (Direction Générale de l’Alimentation),
- la DGCCRF (Direction Générale de la Consommation, de la Concurrence et de la Répression des Fraudes),
- l'ANSES (Agence nationale de sécurité sanitaire de l’alimentation, de l’environnement et du travail) avec le Laboratoire National de Référence des Listeria monocytogenes (LNR Lm),
- l’AFNOR (Association Française de Normalisation) avec la Commission V08B Microbiologie des Aliments,
- plus de 700 laboratoires français, publics ou privés, de biologie médicale ou d’hygiène des aliments.
Au niveau européen
- l’ECDC (European Centre for Prevention and Disease Control) avec le réseau FWD (Food and waterborne diseases and zoonoses),
- le CEN (Comité Européen de Normalisation),
- l’EFSA (European Food Safety Authority)
Au niveau international
- l’OMS (Organisation Mondiale de la Santé),
- le Réseau International des Centres Nationaux de Référence (CNR) des Listeria de l’OMS,
- la FAO (Organisation des Nations Unies pour l’Alimentation et l’Agriculture),
- l’OIE (Office International des Epizooties),
- le CDC (Centers for Disease Control and Prevention, USA).
Activités 2019-2020
En 2019-2020, le Centre National de Référence Listeria (CNRL) a reçu et analysé 5288 isolats de Listeria [2020 : 2039; 2019 : 3249] dans le cadre de ses missions de surveillance dont : 747 isolats [2020 : 346; 2019 : 401] issus de 681 cas humains [2020 : 320; 2019 : 361], 2559 [2020 : 847; 2019 : 1712] isolats d’alertes produits, 769 [2020 : 251; 2019 : 518] isolats d’autocontrôles et 1213 [2020 : 595; 2019 : 618] isolats analysés dans le cadre d’investigations de clusters passés et d’étude de la biodiversité.
La méthode de typage moléculaire des isolats est le core génome MultiLocus Sequence Typing (cgMLST) obtenue à partir du séquençage du génome depuis 2015 au CNRL et devenu technique de première intention pour la surveillance microbiologique de Listeria monocytogenes en France depuis 2017.
En 2019-2020, 247 clusters [2020 : 123; 2019 : 131] ont été détectés par le CNRL et investigués en lien avec SPF et les autres partenaires de la cellule Listeria. Sur les 123 nouveaux clusters [2020 : 54; 2019 : 69], la source de contamination a été identifiée pour 22 d’entre eux, soit 18% [2020 : 6; 2019 : 16].
Le CNRL a aussi participé à la réponse aux alertes européennes à la plateforme EPIS (Epidemic Intelligence Information System) de l’ECDC [2020 : 7; 2019 : 20].
Le CNRL a publié des études de caractérisation de la biodiversité, virulence et résistance de Listeria monocytogenes, afin de mieux comprendre leur association aux différents types d’aliments, et leur pouvoir pathogène. Le CNRL a également publié des études sur la listériose materno-néonatale et les formes rares de listériose (infections endovasculaires et cutanées).
Le CNRL a participé à plusieurs demandes nationales et internationales d’expertises dans le contexte de la surveillance microbiologique et de la recherche sur Listeria.
Mis à jour le 10/02/2022

