Dans un contexte où la résistance de Plasmodium à l’artémisinine, dernier traitement efficace contre le paludisme, devient un obstacle dans la lutte contre la maladie, des chercheurs de l’Institut Pasteur et du CNRS ont résolu la structure tridimensionnelle d’une nouvelle cible thérapeutique. Nommée SUB1, cette protéine joue un rôle essentiel lors de la sortie du parasite des cellules hôtes puis, dans son entrée dans de nouvelles cellules. Ces résultats, publiés dans la revue Nature Communications, le 10 septembre 2014, décryptent le mécanisme d’activation de SUB1, étape initiale engageant le processus de sortie du parasite des cellules hôtes. De plus, les informations structurales permettent d’optimiser le développement d’inhibiteurs de SUB1 dans le but d’identifier de nouveaux candidats antipaludiques.
Communiqué de presse
Paris, le 10 septembre 2014
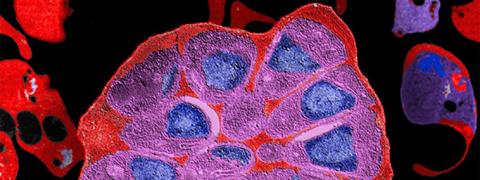
Près de 40% de la population mondiale est exposée au paludisme. Provoqué par les parasites du genre Plasmodium, le paludisme touche chaque année environ 220 millions de personnes et est responsable de près de 660 000 morts. L’émergence récente en Asie du Sud-Est de parasites résistants à l’artémisinine, jusque là dernier antipaludique efficace, constitue une menace pour lutter efficacement contre la maladie.
Après avoir été injecté dans la peau par la piqûre d’un moustique Anophèle, le parasite responsable du paludisme se loge dans les cellules du foie, les hépatocytes. Puis, les parasites vont se multiplier et sortir des hépatocytes, pour infecter les globules rouges du sang.
Les chercheurs de l’équipe de Jean-Christophe Barale (Unité de Biologie et Génétique du Paludisme de l’Institut Pasteur et du CNRS) ont déchiffré le mécanisme de régulation spatio-temporelle de la protéine parasitaire SUB1, laquelle joue un rôle crucial pour initier la sortie des parasites des hépatocytes et des globules rouges. SUB1 active en effet différentes molécules impliquées dans la rupture de la double membrane entourant les parasites intracellulaires, permettant ainsi leur sortie. De plus, SUB1 prépare les parasites libérés, les rendant compétents pour l’étape suivante, à savoir l’invasion de nouveaux globules rouges. Ainsi se perpétuent les formes sanguines de Plasmodium, responsables des symptômes du paludisme, dont le plus connu, la forte fièvre, est justement associé à la phase de sortie du parasite des globules rouges.
SUB1 : une cible thérapeutique et préventive innovante
SUB1 est une cible thérapeutique innovante : bloquer son action aux stades hépatiques préviendrait l’apparition des formes sanguines, donc des symptômes du paludisme. Bloquer SUB1 alors qu’une phase sanguine est déjà engagée la stopperait. Ainsi, un inhibiteur de SUB1 aurait une double action : préventive, via son action dès les stades hépatiques, et thérapeutique, via son action sur les stades sanguins de Plasmodium.
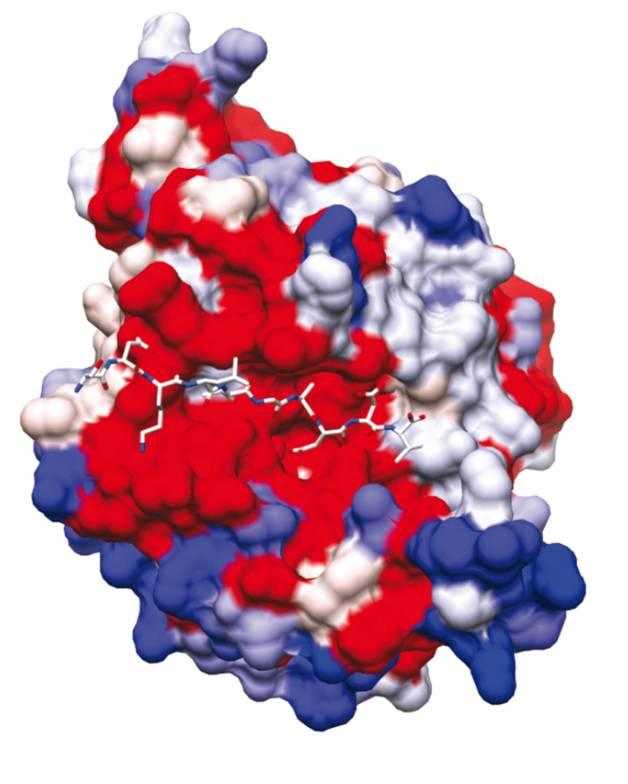
Dans cette étude, afin d’affiner la recherche de molécules qui pourraient bloquer ce verrou de sortie des parasites que représente SUB1, les chercheurs des équipes de Pedro Alzari et de Jean-Christophe Barale (Unité de Microbiologie Structurale et unité de Biologie et Génétique du Paludisme, de l’Institut Pasteur et du CNRS) sont parvenus à résoudre la structure tridimensionnelle de SUB1 telle qu’elle existe juste avant la libération des parasites.
Le rôle de SUB1 est de couper à des sites très précis différentes protéines, ce qui les active, leur permettant d’aller rompre la double membrane entourant le parasite. A l’image d’une serrure et d’une clé, SUB1 accueille spécifiquement ces protéines dans son sillon catalytique, où elles seront coupées et activées.
Les chercheurs de l’Institut Pasteur et du CNRS ont montré que la serrure (sillon catalytique) de SUB1 est naturellement protégée par une ceinture, ce qui rend SUB1 inopérante. Ils sont parvenus à élucider le stratagème sophistiqué permettant à SUB1 d’ôter cette ceinture, libérant alors le sillon catalytique/serrure au moment précis où les parasites sont mûrs, prêts à sortir de la cellule pour en envahir de nouvelles. Ils ont réussi à montrer que c’est la forte augmentation du taux de calcium dans la cellule qui est responsable de la libération de la serrure de SUB1, ce qui l’active, initiant ainsi le processus de sortie des parasites. Le mécanisme d’un des verrous de la libération des parasites mûrs est donc élucidé.
De plus, la structure et la composition du sillon catalytique de SUB1 ont été très précisément décrites : cela permet de mieux prédire la forme que doivent avoir les composés chimiques pour s’y loger, bloquer la serrure SUB1 et empêcher la sortie des parasites. Ces résultats, issus d’une approche multidisciplinaire, représentent une étape cruciale dans la recherche d’inhibiteurs de SUB1 qui pourraient être à l’origine d’une nouvelle génération d’antipaludiques.
Illustrations :
Bandeau : Globule rouge parasité par Plasmodium falciparum, © Institut Pasteur / Biologie des Interactions Hôte-Parasite
Vignette : Structure tridimensionnelle de SUB1. En rouge au centre : le sillon catalytique. © Jean-Christophe Barale, Institut Pasteur
Cette étude a reçu un financement de l’Agence Nationale de la Recherche, de l'Institut Carnot-Pasteur Maladies Infectieuses, du Fond dédié : Combattre les maladies parasitaires (Sanofi / ministère de la Recherche) et du ministère de la Défense (DGA).
Source
A novel Plasmodium-specific prodomain fold regulates the malaria drug target SUB1 subtilase, Nature Communications, 10 septembre 2014. DOI : NCOMMS5833
David Giganti(1*), Anthony Bouillon(2*), Lina Tawk(2), Fabienne Robert(2), Mariano Martinez(1), Elodie Crublet(3), Patrick Weber(3), Christine Girard-Blanc(3), Stéphane Petres(3), Ahmed Haouz(3), Jean-François Hernandez(4), Odile Mercereau-Puijalon(2), Pedro M. Alzari(1,3)& Jean-Christophe Barale(2).
(1) Institut Pasteur, Unité de Microbiologie Structurale, Département de Biologie Structurale et Chimie & CNRS UMR 3528, F-75015 Paris, France
(2) Institut Pasteur, Unité d’Immunologie Moléculaires des Parasites, Département de Parasitologie et de Mycologie & CNRS URA 2581, F-75015 Paris, France
(3) Institut Pasteur, Proteopole & CNRS UMR 3528, F-75015 Paris, France
(4) Institut des Biomolécules Max Mousseron, UMR5247, CNRS, Universités Montpellier 1 & 2, Faculté de Pharmacie 15 avenue Charles Flahault, 34093 Montpellier cedex 5, France
*Ces auteurs ont contribué équitablement à ce travail.