En prenant pour modèle l’épidémie de Chikungunya de 2005-2006, des chercheurs de l’Institut Pasteur, du CNRS et de l’Institut Pasteur du Cambodge sont parvenus à mettre au point une méthode capable de prédire les mutations virales les plus susceptibles d’émerger à court terme, et présentant un fort potentiel épidémique. Ces travaux ouvrent des perspectives nouvelles, tant pour la surveillance des épidémies que pour les recherches vaccinales menées sur de nombreux virus. Ces résultats sont publiés dans la revue Cell Host & Microbe.
Communiqué de presse
Paris, le 12 juin 2014
Les mécanismes moléculaires de réplication et de multiplication des virus à ARN génèrent parfois des erreurs – ou mutations – dans leur code génétique, aboutissant à la production de virus légèrement différents les uns des autres. Ces événements sont fréquents, et la population virale d’une même espèce est en réalité constituée d’un nuage de virus, aux mutations variées. Certaines d’entre elles confèrent au virus qui en est porteur un avantage sélectif : pour les arbovirus qui circulent entre insectes et vertébrés par exemple, elles favorisent sa multiplication chez le moustique vecteur ou chez l’hôte mammifère qu’il infecte, et le rendent donc davantage apte à se répandre. La souche virale en question possède alors le potentiel pour déclencher une épidémie d’ampleur.
Des chercheurs de l’unité de Marco Vignuzzi (Institut Pasteur/CNRS) sont parvenus pour la première fois à développer une méthode capable de prédire, dans une population d’arbovirus, comme ceux de la dengue, du chikungunya ou de la fièvre jaune, les mutations à fort potentiel épidémique les plus susceptibles d’émerger. Cette méthode repose sur l’observation, dans les conditions naturelles, sans évolution expérimentale forcée, des événements mutationnels chez le virus, au cours de son cycle chez le moustique et l’hôte mammifère.
Les scientifiques ont dans un premier temps mis au point et vérifié la validité de leur démarche en s’appuyant sur le cas réel de l’épidémie de Chikungunya de 2005-2006 dans l’Océan Indien. Ils ont pour cela infecté, avec la souche sauvage pré-épidémique d’origine, les deux espèces de moustiques capables de transmettre le virus : Aedes aegypti, le vecteur traditionnel, et Aedes albopictus, le moustique tigre. Ils ont ensuite laissé le virus se développer sur une longue période (14 jours), correspondant à plusieurs cycles de réplication, puis procédé à un séquençage à haut débit du génome des virus prélevés dans différents sites dont la salive de chaque insecte infecté. Les résultats ont alors montré l’apparition, de manière majoritaire dans la salive, de la même mutation dans la protéine d’enveloppe E1 (A226V) qui en 2005 a été à l’origine de la souche épidémique. Par ailleurs, comme au cours de l’épidémie réelle, c’est le moustique Ae. albopictus qui s’est finalement révélé le plus apte à développer la souche mutée épidémique.
Les scientifiques ont alors appliqué leur méthode pour simuler les événements naturels possibles lors d’une prochaine épidémie de Chikungunya. Ils ont ainsi effectué en laboratoire un cycle de transmission complet (moustique I – souris - moustique II), en infectant la première génération de moustiques avec la souche épidémique mutée apparue en 2005-2006. A l’issue de cette phase d’infection, deux mutations dans la protéine d’enveloppe E1 (V80I et A129V) sont apparues dans la salive des moustiques I, restant encore minoritaires par rapport à la souche de départ. Mais après le passage chez l’hôte mammifère et les moustiques II, les souches virales qui en étaient porteuses ont supplanté toutes les autres, y compris la souche épidémique, pointant ainsi, après seulement un cycle de transmission, le fort potentiel émergent de ces nouvelles souches mutées. Les analyses in vitro suggèrent que les avantages sélectifs de ces deux mutations pourraient être liés à une capacité accrue du virus à fusionner avec les membranes de cellules cibles de l’infection et une meilleure stabilité thermique dans les milieux extracellulaires comme la salive ou le sang des mammifères.
Outre la mise en évidence de ces deux mutations probables du virus Chikungunya, les travaux des chercheurs fournissent aujourd’hui un outil permettant d’identifier plus en amont les mutations au potentiel épidémique important, pour les arbovirus comme pour d’autres virus , et notamment les virus à transmission vectorielle. Cette découverte offre donc désormais la possibilité de cibler et d’améliorer la surveillance de ces populations virales, en routine et au cours d’épisodes épidémiques. En permettant d’inclure dans les compositions vaccinales les souches virales potentielles identifiées, elle constitue également un atout précieux pour la mise au point de vaccins et la lutte contre ces virus.
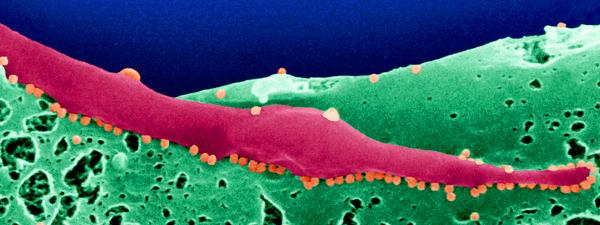
Illustration : virus du Chikungunya (en orange) bourgeonnant à la surface de cellules infectées de moustique Aedes albopictus. ©Institut Pasteur
Cette étude a été financée par l’European Research Council, la fondation Bill et Melinda Gates, le programme Investissement d’avenir LabEx IBEID et le Haut-Conseil franco-israëlien pour la science et la technologie.
Source
Emergence and Transmission of Arbovirus Evolutionary Intermediates with Epidemic Potential, Cell Host & Microbe, 11 juin 2014.
Kenneth A. Stapleford (1), Lark L. Coffey (2), Sreyrath Lay (3), Antonio V. Bordería (1), Veasna Duong (3), Ofer Isakov (4), Kathryn Rozen-Gagnon (1), Camilo Arias-Goeta (5), Hervé Blanc (1), Stéphanie Beaucourt (1), Türkan Haliloğlu (6), Christine Schmitt (7), Isabelle Bonne (7), Nir Ben-Tal (8), Noam Shomron (4), Anna-Bella Failloux (5), Philippe Buchy (3) and Marco Vignuzzi (1).
(1) Unité Populations virales et pathogénèse, Institut Pasteur, Centre National de la Recherche Scientifique UMR 3569, 28 rue du Dr Roux,75724 Paris Cedex 15, France
(2) Center for Vectorborne Diseases, Department of Pathology, Microbiology and Immunology, School of Veterinary Medicine, University of California, Davis, One Shields Avenue, 5327 VM3A, Davis, CA 95616, USA
(3) Unité de Virologie, Institut Pasteur in Cambodia 5, Monivong Boulevard, PO Box 983, Phnom Penh, Cambodia
(4) Sackler Faculty of Medicine, Tel Aviv University, Tel Aviv 69978, Israel
(5) Laboratoire Arbovirus et insectes vecteurs, Institut Pasteur, 25 rue du Dr Roux, 75724 Paris Cedex 15, France
(6) Department of Chemical Engineering and Polymer Research Center, Boğaziçi University, Bebek 34342, Istanbul, Turkey
(7) Ultrastructural Microscopy Platform, Institut Pasteur, 28 rue du Dr Roux, 75724 Paris Cedex 15, France
(8) Department of Biochemistry and Molecular Biology, George S. Wise Faculty of Life Sciences, Tel Aviv University, Tel Aviv 69978, Israel