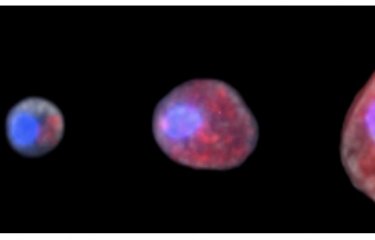
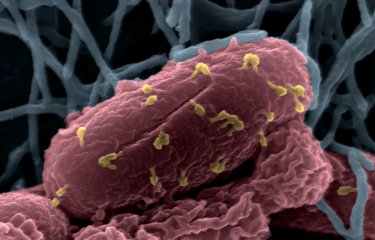
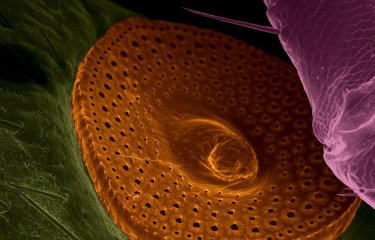
La leishmaniose est une maladie humaine et animale causée par les parasites Leishmania. Elle touche 12 millions de personnes et est endémique dans une centaine de pays. Elle émerge à présent en Europe en raison de changements climatiques et de déplacements massifs de population. Le parasite Leishmania est connu pour s’adapter rapidement à de nouveaux environnements, avec des conséquences importantes sur l’évolution de la maladie. L’Union européenne a, par conséquent, reconnu la leishmaniose comme une menace émergente pour la santé publique. Dans un article publié par la revue Nature Ecology & Evolution, des chercheurs de l’Institut Pasteur à Paris et du Centre de régulation génomique (CRG) de Barcelone, en collaboration avec des équipes de l’Institut de médecine tropicale (IMT) d’Anvers et de l’Université de Montpellier, apportent la preuve que l’adaptation des leishmanies résulte d’amplifications chromosomiques fréquentes et réversibles. Ces variations, appelées aneuploïdies, sont semblables à celles constatées dans de nombreux types de cancers. Ces découvertes représentent une avancée considérable dans la compréhension de l’infection humaine à Leishmania, notamment en termes de pharmaco-résistance des parasites, de pathogénicité et de tropisme tissulaire. Cette perspective inédite de l’instabilité génomique chez les leishmanies devrait ouvrir la voie à l’identification des mécanismes de résistance de ces parasites aux médicaments dans des conditions cliniquement pertinentes et contribuer à identifier des biomarqueurs ayant une valeur de diagnostic et de pronostic.
La leishmaniose compte parmi les cinq principales maladies parasitaires dans le monde. On estime à 350 millions le nombre de personnes exposées au risque d’infection. Les manifestations cliniques de cette maladie sont variées – des formes cutanées défigurantes aux formes viscérales mortelles – et dépendent des différentes espèces parasitaires responsables de l’infection. Les parasites unicellulaires Leishmania s’adaptent à une multitude d’hôtes. Ils se développent sous leur forme extracellulaire dans des insectes appelés phlébotomes qui les transmettent ensuite à divers hôtes vertébrés, tels que les rongeurs, les chiens et les humains. Chez ces derniers, ils évoluent et prolifèrent au sein des cellules immunitaires, principalement dans les macrophages, causant des pathologies graves et parfois mortelles.
Compte tenu de son incidence mondiale, la leishmaniose est l’une des maladies les plus négligées. Aucun vaccin humain n’existe, et seuls quelques traitements sont disponibles, lesquels présentent des limitations notables en termes d’administration, de toxicité ou de coût. De plus, la biologie des leishmanies se caractérise par la capacité de ces parasites à s’adapter aux diverses fluctuations rencontrées à l’intérieur de l’hôte humain, notamment en réponse aux interventions pharmacologiques, ce qui n’est pas sans conséquence sur la progression de la maladie, comme démontré par l’émergence d’isolats cliniques résistants aux médicaments.
Pour identifier les mécanismes génétiques qui conduisent à l’adaptation génomique de Leishmania, des chercheurs de l’Institut Pasteur à Paris et du Centre de régulation génomique (CRG) de Barcelone, en collaboration avec des équipes de l’Institut de médecine tropicale (IMT) d’Anvers et de l’Université de Montpellier, ont développé des techniques inédites de génomique comparative s’appuyant sur des analyses in silico et sur le séquençage de nouvelle génération (NGS). Leur travail montre pour la première fois l’existence d’un rapport entre l’évolution du nombre de copies chromosomiques et la sélection de nouveaux allèles nécessaires à la survie des leishmanies.
L’aspect le plus surprenant de leur recherche réside dans l’observation de l’aptitude de Leishmania à allier sélection d’allèles et maintien d’une grande diversité génétique. Ces deux processus devraient normalement s’exclure mutuellement, et l’on s’attendrait plutôt à ce qu’un parasite soumis à une forte sélection perde rapidement sa diversité génétique. C’est en tout cas ce qui se passerait si les duplications chromosomiques très fréquentes ne rendaient possible la coexistence d’un processus de sélection avec un maintien significatif de la diversité génétique. En effet, les duplications fréquentes permettent à une même combinaison d’allèles d’être sélectionnée simultanément dans des individus génétiquement différents.
À la tête de l’unité Parasitologie moléculaire et signalisation à l’Institut Pasteur à Paris, Gérald Späth, qui a dirigé la partie expérimentale de cette étude, commente : « La recherche fondamentale, appliquée et clinique sur Leishmania est presque exclusivement menée à l’aide de parasites maintenus en culture à long terme. Notre étude montre que le génome de ces parasites évolue très vite en laboratoire. Cet aspect doit être pris en compte lors de l’étude de leur biologie et de la recherche de nouveaux biomarqueurs, médicaments ou candidats vaccins. À l’avenir, les recherches sur les leishmanies doivent s’appuyer sur une méthode plus intégrative, qui considère les interactions génétiques complexes entre le parasite, l’hôte vertébré et l’insecte vecteur, et être réalisées dans des conditions cliniquement pertinentes, par exemple à l’aide de parasites en culture à court terme ou par séquençage direct des tissus. »
Directeur de recherche au Centre de régulation génomique (CRG) de Barcelone, Cédric Notredame, qui a dirigé la partie « génomique comparative » de cette étude, explique : « L’idée que l’amplification génétique est une voie d’adaptation n’est pas nouvelle, mais notre travail montre que chez Leishmania, l’évolution a intensifié ce mécanisme au point d’en faire une partie essentielle du cycle de vie des parasites. Un nombre croissant de travaux révèlent que des aneuploïdies tout aussi complexes jouent un rôle dans certains cancers, et nous pensons que le recueil rapide de données génomiques NGS associé à des techniques in silico inédites, comme celles développées dans notre étude, permettra bientôt de mieux comprendre les relations entre aneuploïdie et sélection d’allèles. »
Le Professeur Jean-Claude Dujardin, de l’IMT, souligne : « Il nous a fallu plus de cinq ans pour collecter un jeu de données de séquençage sans précédent à partir d’isolats cliniques du sous-continent indien et d’en publier une première analyse l’année dernière. En rendant publiques toutes les données de séquençage, la science moderne donne libre cours à de nouvelles collaborations et des études comme la nôtre. Elle a également permis de créer une alliance inédite entre l’IMT, l’Institut Pasteur et le CRG dans la lutte contre cette maladie infectieuse. »
Les résultats de cette étude originale s’avèrent hautement pertinents pour d’autres maladies humaines reposant sur l’instabilité génomique, telles que les infections fongiques et les cancers. Ils offrent aussi des perspectives inédites de découverte de médicaments anti-leishmaniose via des stratégies ciblant l’hôte, c’est-à-dire la dépendance métabolique des parasites vis-à-vis de la cellule hôte, et empêchant ainsi l’évolution adaptative des parasites résistants aux médicaments.
Fort des résultats publiés dans cet article et animé par la volonté d’étudier la biologie et l’épidémiologie de Leishmania dans un contexte cliniquement plus pertinent, le Dr Gérald Späth a créé le consortium international « LeiSHield » (www.leishield.org), qui coordonne l’effort collectif des équipes partenaires, notamment du Réseau international des instituts Pasteur. Ce consortium a reçu une aide financière initiale de la direction internationale de l’Institut Pasteur et bénéficie désormais d’une subvention d’1,7 million d’euros du programme européen Horizon 2020.
Source
Haplotype selection as an adaptive mechanism in the protozoan pathogen Leishmania donovani, Nature Ecology & Evolution, 6 novembre 2017. DOI : 10.1038/s41559-017-0361-x
Pablo Prieto Barja (1,6,$), Pascale Pescher (2,$), Giovanni Bussotti (3), Franck Dumetz (4), Hideo Imamura (4), Darek Kedra (1), Malgorzata Domagalska (4), Victor Chaumeau (5), Heinz Himmelbauer (1,7), Michel Pages (5), Yvon Sterkers (5), Jean-Claude Dujardin (4,8), Cedric Notredame (1,6,*) & Gerald Frank Späth (2,*)
(1) Centre for Genomic Regulation (CRG), The Barcelona Institute of Science and Technology, Barcelona, Spain;
(2) Institut Pasteur, INSERM U1201, Unité de Parasitologie moléculaire et Signalisation, Paris, France;
(3) HUB de Bioinformatique et Biostatistiques, Centre de Bioinformatique, Biostatistique et Biologie Intégrative (C3BI), Institut Pasteur, Paris, France;
(4) Institute of Tropical Medicine, Molecular Parasitology Unit, Antwerpen, Belgium;
(5) University Montpellier, Faculty of Medicine, Laboratory of Parasitology–Mycology, CNRS 5290, IRD 224, University Montpellier (UMR “MiVEGEC”) and Centre Hospitalier Universitaire, Montpellier, France;
(6) Universitat Pompeu Fabra (UPF), Barcelona, Spain;
(7) Institute of Biotechnology Muthgasse 18 1190 Vienna;
(8) Universiteit Antwerpen, Department of Biomedical Sciences, Belgium
* Corresponding authors