Des chercheurs de l’Institut Pasteur et de son réseau international, du Wellcome Trust Sanger Institute (Cambridge, Royaume Uni) et de nombreuses institutions internationales viennent de publier une étude d’une envergure exceptionnelle, retraçant l’histoire du bacille de la dysenterie épidémique, l’un des pires fléaux qu’ait connu l’humanité aux cours des XVIIIe et XIXe siècles. Cette vaste enquête scientifique permet pour la première fois d’établir les liens entre les différents foyers épidémiques au cours du temps. Elle révèle notamment que le pathogène sévissant actuellement en Afrique et en Asie, probablement d’origine européenne, a été transmis d’un continent à l’autre au travers des flux migratoires et des opérations militaires. Elle met également en évidence l’apparition de son inéluctable résistance aux antibiotiques. Cette étude est publiée le 21 mars dans la revue Nature Microbiology.

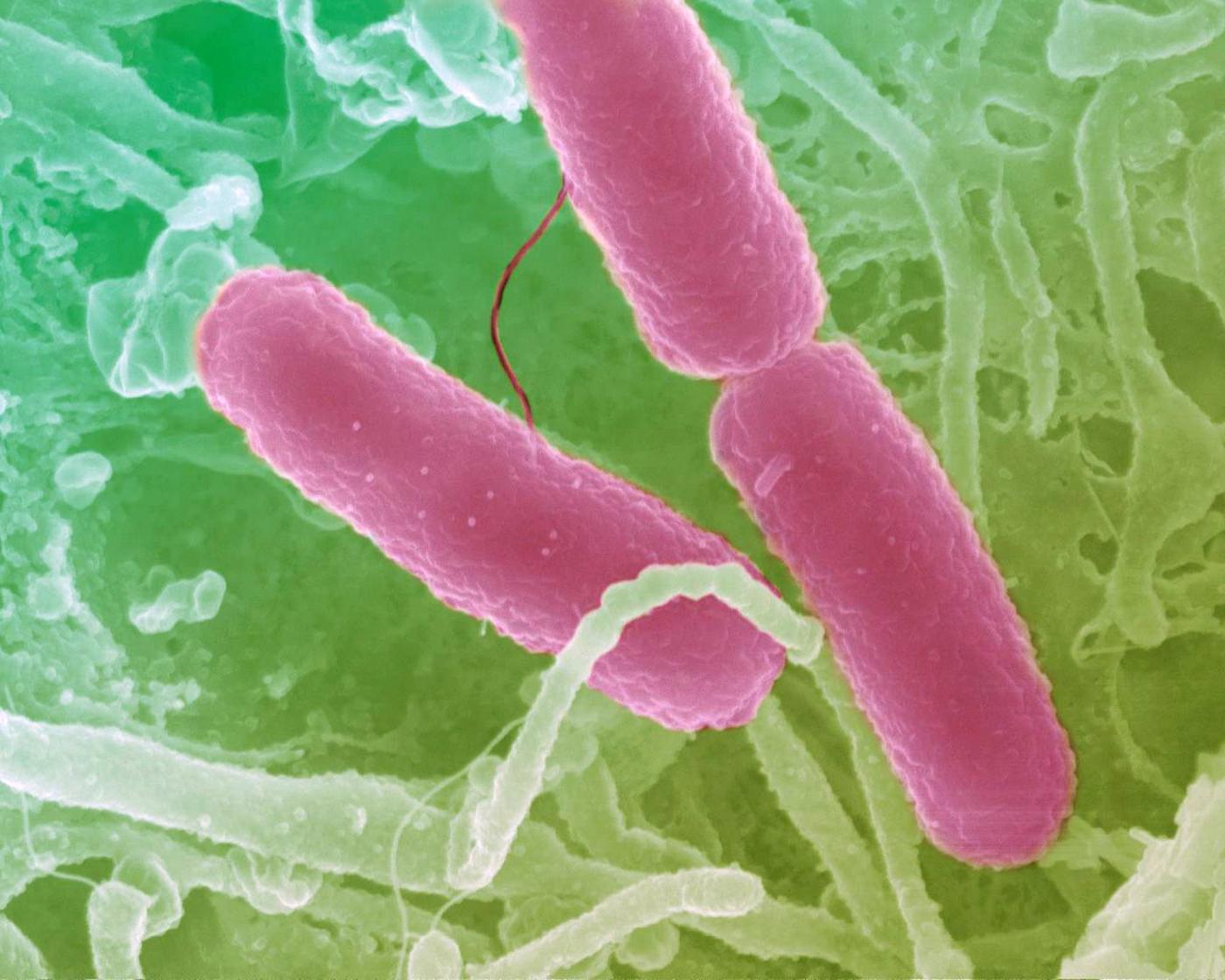
Shigella, agent de la dysenterie bacillaire, image colorisée. © Institut Pasteur
L’agent de la dysenterie épidémique, la bactérie Shigella dysenteriae de type 1, a été isolé au Japon en 1897 par le Dr Kyoshi Shiga au cours d’une vaste épidémie de diarrhée sanglante ayant provoqué 20 000 morts en l’espace de six mois. Depuis, les souches ont été isolées aux quatre coins du monde, sans qu’il ne soit possible de comprendre l’origine des épidémies auxquelles elles ont donné naissance, et leurs liens qui les unissent.
Pour répondre à cette question, des chercheurs, menés par François-Xavier Weill (unité des Bactéries pathogènes entériques, Institut Pasteur) et Nicholas Thomson (Bacterial Genomics and Evolution, Wellcome Trust Sanger Institute) ont entrepris une enquête génomique sans précédent : faisant appel aux nouvelles technologies de séquençage du génome bactérien et de bioinformatique, ils ont analysé plus de 330 souches de Shigella dysenteriae type 1 isolées de 1915 – chez des soldats du corps expéditionnaire des Dardanelles – à 2011 et collectées par 35 instituts internationaux dans 66 pays.
Contrairement aux idées reçues, l’étude révèle notamment que le pathogène Shigella dysenteriae sévissant actuellement en Afrique et en Asie est certainement d’origine européenne. Les lignées les plus ancestrales ayant circulé en Europe au cours de la première partie du XXe siècle, les chercheurs estiment que leur ancêtre commun existait déjà dans les années 1740, et qu’il pourrait être le responsable des violentes épidémies de diarrhées sanglantes décrites au cours du XVIIIe et XIXe siècle en Europe de l’Ouest et du Nord, bien avant l’avènement des travaux de Louis Pasteur et de la microbiologie. Ces épidémies de dysenterie tuèrent plus de 200 000 personnes en France en 1738-1742 et 1779 notamment, et 35 000 en Suède en 1770-1775. Au XIXe siècle, l’Irlande a également été sévèrement touchée lors de la grande famine de 1846-1849 et la France de nouveau lors de la guerre de 1870.
Un pathogène qui profite de l’expansion européenne
En identifiant différentes lignées génétiques, les scientifiques ont pu retracer le cheminement de la bactérie à l’échelle mondiale au cours du temps. En l’espace de moins de 20 ans, entre 1889 et 1903, S. dysenteriae, partant d’Europe, s’est d’abord implantée en Amérique, en Afrique et en Asie. Cette dissémination a probablement bénéficié des mouvements de populations dues à l’émigration européenne en Amérique ainsi qu’à la colonisation de différents territoires en Afrique et en Asie. L’ensemble a en outre été certainement favorisé par la marine à vapeur et l’ouverture du Canal de Suez en 1869.
Au XXe siècle, la bactérie est une nouvelle fois identifiée en Europe lors du premier conflit mondial, en particulier lors de l’expédition des Dardanelles (1915-1916), où elle contribua grandement à la défaite des troupes alliées. Elle est également retrouvée en Europe centrale, lors de la seconde guerre mondiale, avant de disparaître du continent.
En revanche, elle continue son expansion en Asie, en Afrique et en Amérique centrale sous formes de flambées épidémiques violentes (500 000 cas pour l’épidémie d’Amérique centrale de 1969-1972). Le foyer du sous-continent Indien (Inde et Bengladesh) deviendra par la suite le plus actif tout au long du XXe siècle et sera la source de plusieurs vagues épidémiques vers l’Afrique et l’Asie du Sud Est.
En l’espace de 25 ans (1965-1990), 99% des souches sont devenues résistantes aux antibiotiques. L’étude de la collection, dont les premières bactéries ont été isolées bien avant l’utilisation des antibiotiques chez l’homme, a également permis de dater précisément l’apparition des premières résistances aux antibiotiques au milieu des années 1960 en Asie et en Amérique. La bactérie a ensuite accumulé des gènes de résistance vis-à-vis de la plupart des classes d’antibiotiques, et depuis les années 1990, moins de 1% des souches bactériennes restent sensibles aux antibiotiques. Le bacille de Shiga circulant dans les mêmes zones que ces gènes de résistance, les chercheurs estiment, de manière préoccupante, que l’apparition de bactéries résistantes aux dernières classes d’antibiotiques est aujourd’hui inéluctable. Cependant, ce pessimisme peut être tempéré par une plus grande rareté de l’infection depuis 2010, y compris en Asie du Sud et en Afrique. Mais si la bactérie n’est plus désormais isolée lors de vagues épidémiques, elle est toujours retrouvée lors d’enquêtes de terrain comme celle réalisée par Médecins sans Frontières au Niger en 2011 chez des enfants diarrhéiques. Pour le Dr François-Xavier Weill, le bacille de Shiga circule donc encore a bas bruit et pourrait être de nouveau responsable d’épidémies s’il devait rencontrer des circonstances propices, comme un rassemblement important de personnes sans accès à l’eau potable ni traitement des déchets humains. Selon lui, cette étude souligne la nécessité d’un vaccin efficace, qui s’impose comme un moyen de contrôle plus sûr à l’avenir que l’utilisation des antibiotiques.
Cette étude a bénéficié du financement de l’Institut Pasteur, du Réseau international des Instituts Pasteur, de l’InVS, du Labex IBEID, de la Fontation Le Roch Les Mousquetaires, ainsi que du Wellcome Trust.
Source
Global phylogeography and evolutionary history of Shigella dysenteriae type 1', Nature Microbiology, 21 mars 2016.
Elisabeth Njamkepo (1)†, Nizar Fawal (1)†, Alicia Tran-Dien (1)†, Jane Hawkey (2,3,4), Nancy Strockbine (5), Claire Jenkins (6), Kaisar A. Talukder (7), Raymond Bercion (8,9), Konstantin Kuleshov (10), Renáta Kolínská (11), Julie E. Russell (12), Lidia Kaftyreva (13), Marie Accou-Demartin (1), Andreas Karas (14), Olivier Vandenberg (15,16), Alison E. Mather (17,18), Carl J. Mason (19), Andrew J. Thandavarayan Ramamurthy (20), Chantal Bizet (21), Andrzej Gamian (22), Isabelle Carle (1), Amy Gassama Sow (9), Christiane Bouchier (23), Astrid Louise Wester (24), Monique Lejay-Collin (1), Marie-Christine Fonkoua (25), Simon Le Hello (1), Martin J. Blaser (26), Cecilia Jernberg (27), Corinne Ruckly (1), Audrey Mérens (28), Anne-Laure Page (29), Martin Aslett (17), Peter Roggentin (30), Angelika Fruth (31), Erick Denamur (32), Malabi Venkatesan (33), Hervé Bercovier (34), Ladaporn Bodhidatta (19), Chien-Shun Chiou (35), Dominique Clermont (21), Bianca Colonna (36), Svetlana Egorova (13), Gururaja P. Pazhani (20), Analia V. Ezernitchi (37), Ghislaine Guigon (38), Simon R. Harris (17), Hidemasa Izumiya (39), Agnieszka Korzeniowska-Kowal (22), Anna Lutyńska (40), Malika Gouali (1), Francine Grimont (1), Céline Langendorf (29), Monika Marejková (41), Lorea A.M. Peterson (42), Guillermo Perez-Perez (26), Antoinette Ngandjio (25), Alexander Podkolzin (10), Erika Souche (43), Mariia Makarova (13), German A. Shipulin (10), Changyun Ye (44), Helena Žemličková (11,45), Mária Herpay (46), Patrick A. D. Grimont (1), Julian Parkhill (17), Philippe Sansonetti (47), Kathryn E. Holt (2,3), Sylvain Brisse (38,48,49), Nicholas R. Thomson (17,50) and François-Xavier Weill (1,17)*
(1) Institut Pasteur, Enteric Bacterial Pathogens Unit, 75724 Paris Cedex 15, France.
(2) Centre for Systems Genomics, University of Melbourne, Parkville, Victoria 3010, Australia.
(3) Department of Biochemistry and Molecular Biology, Bio21 Molecular Science and Biotechnology Institute, University of Melbourne, Parkville, Victoria 3010, Australia.
(4) School of Agriculture and Veterinary Science, University of Melbourne, Parkville, Victoria 3010, Australia.
(5) Centers for Disease Control and Prevention, Escherichia and Shigella Reference Unit, Atlanta, Georgia 30333, USA.
(6) Public Health England, Gastrointestinal Bacteria Reference Unit, Colindale NW9 5HT, UK.
(7) icddr,b, Enteric and Food Microbiology Laboratory, Dhaka 1212, Bangladesh.
(8) Institut Pasteur in Bangui, BP 923, Bangui, Central African Republic.
(9) Institut Pasteur in Dakar, BP 220, Dakar, Senegal.
(10) Federal Budget Institute of Science, Central Research Institute for Epidemiology, Moscow 111123, Russia.
(11) Czech National Collection of Type Cultures (CNCTC), National Institute of Public Health, Prague 10, Czech Republic.
(12) Public Health England, National Collection of Type Cultures, Porton Down SP4 0JG, UK.
(13) Pasteur Institute of St Petersburg, St Petersburg, 197101, Russia.
(14) Department of Medical Microbiology, University of KwaZulu-Natal, Durban 4041, South Africa.
(15) Department of Microbiology, LHUB-ULB, Brussels University Hospital Laboratory, 1000 Brussels, Belgium.
(16) Environmental Health Research Centre, Public Health School, Université Libre de Bruxelles, 1070 Brussels, Belgium.
(17) Wellcome Trust Sanger Institute, Cambridge CB10 1SA, UK.
(18) Department of Veterinary Medicine, University of Cambridge, Cambridge CB3 0ES, UK.
(19) Armed Forces Research Institute of Medical Sciences (AFRIMS), Bangkok 10400, Thailand.
(20) National Institute of Cholera and Enteric Diseases (NICED), Kolkata, West Bengal 700010, India.
(21) Institut Pasteur, Institut Pasteur Collection (CIP), 75724 Paris Cedex 15, France.
(22) Polish Collection of Microorganisms, Institute of Immunology and Experimental Therapy, 53-114 Wroclaw, Poland.
(23) Institut Pasteur, Genomics Platform (PF1), 75724 Paris Cedex 15, France.
(24) Department of Foodborne Infections, Norwegian Institute of Public Health, Nydalen 0403, Oslo, Norway.
(25) Pasteur Centre in Cameroon, BP 1274, Yaoundé, Cameroon.
(26) Departments of Medicine and Microbiology, New York University Langone Medical Center, New York, New York 10016, USA.
(27) Public Health Agency of Sweden, 17182 Solna, Sweden.
(28) Biology Department and Infection Control Unit, Bégin Military Hospital, 94160 Saint-Mandé, France.
(29) Epicentre, 75011 Paris, France.
(30) Institut für Hygiene und Umwelt, 20539 Hamburg, Germany.
(31) Division of Enteropathogenic Bacteria and Legionella, Robert Koch Institut, 38855 Wernigerode, Germany.
(32) INSERM, IAME, UMR 1137, Univ. Paris Diderot, IAME, UMR 1137, Sorbonne Paris Cité, 75018 Paris, France.
(33) Bacterial Diseases Branch, Walter Reed Army Institute of Research, Silver Spring, Maryland 20910, USA.
(34) Faculty of Medicine, Hebrew University of Jerusalem, Jerusalem 91120, Israel.
(35) Center of Research and Diagnostics, Centers for Disease Control, Taichung 40855, Taiwan.
(36) Istituto Pasteur-Fondazione Cenci Bolognetti, Dipartimento di Biologia e Biotecnologie C Darwin, Sapienza Università di Roma, 00185, Rome, Italy.
(37) Central Laboratories, Ministry of Health, Jerusalem 91342, Israel.
(38) Institut Pasteur, Genotyping of Pathogens and Public Health Platform, 75724 Paris Cedex 15, France.
(39) Department of Bacteriology I, National Institute of Infectious Diseases, Tokyo, 162-8640, Japan.
(40) Department of Sera and Vaccines Evaluation, National Institute of Public Health–National Institute of Hygiene, 00-791 Warsaw, Poland.
(41) National Reference Laboratory for E. coli and Shigella, National Institute of Public Health, Prague 10, Czech Republic.
(42) National Microbiology Laboratory, Public Health Agency of Canada, Winnipeg, Manitoba R3E 3R2, Canada.
(43) Institut Pasteur, Bioinformatics platform, 75724 Paris Cedex 15, France.
(44) State Key Laboratory of Infectious Disease Prevention and Control, National Institute for Communicable Disease Control and Prevention, China CDC, Beijing 102206, China.
(45) Department of Clinical Microbiology, Faculty of Medicine and University Hospital, Charles University, 500 05, Hradec Kralove, Czech Republic.
(46) Hungarian National Collection of Medical Bacteria, National Center for Epidemiology, H-1097 Budapest, Hungary.
(47) Institut Pasteur, Molecular Microbial Pathogenesis Unit, 75724 Paris Cedex 15, France.
(48) Institut Pasteur, Microbial Evolutionary Genomics Unit, 75724 Paris Cedex 15, France.
(49) CNRS, UMR 3525, 75015, Paris, France.
(50) London School of Hygiene and Tropical Medicine, London WC1E 7HT, UK.
†These authors contributed equally to this work.