- EN
- FR
- EN
- FR

Activités du CNR des Escherichia coli, Shigella, Salmonella
Avant tout envoi, consulter la rubrique « Envoyer une souche »
EXPERTISES
Typage des souches de Escherichia coli, Shigella et Salmonella envoyées par les laboratoires d’analyses de biologie médicale privés (LABM) et publics (hospitaliers) dans le contexte de détection et d’investigation microbiologique de tout phénomène anormal : épidémies, cas groupés, cas cliniques, etc….
Techniques utilisées
Pour les E. coli – Shigella
- CNR associé à l’hôpital Robert Debré de Paris : Recherche des facteurs de pathogénicité des E. coli entérohémorragiques (Stx1, Stx2, eae, ehxA) à partir d’un prélèvement de selles ou d’une souche de E. coli isolée.
- CNR-ESS à l’Institut Pasteur de Paris : séquençage complet du génome des Shigella/EIEC et des STEC isolés au CNR-LA. Les techniques conventionnelles de bactériologie classiques sont toujours présentes au CNR-ESS mais sont utilisées en seconde intention après séquençage complet du génome.
Pour les Salmonella
Chaque souche reçue est identifiée par séquençage complet du génome. Du fait du délai d’obtention et d’analyse des séquences, ou devant des atypies ou des génotypes nouveaux certaines souches sont identifiées par l’étude des caractères biochimiques conventionnels et sérotypées par agglutination à l’aide de sérums spécifiques. Nous utilisons les formules antigéniques du schéma de White-Kauffmann-Le Minor
Volume moyen d’activité
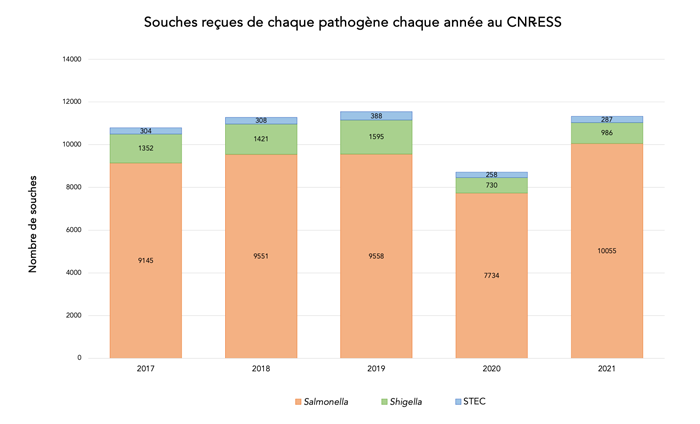
Au cours du dernier mandat (2017 et 2021), le nombre de souches reçues s’est maintenu, sauf en 2020, année de la pandémie à SARS-Cov-2. Toutes les souches de STEC, Shigella et Salmonella reçues au CNR-ESS ont été analysées par WGS.
SURVEILLANCE
L’analyse des activités du CNR Escherichia coli, Shigella, Salmonella permet d’établir des tendances épidémiologiques et d’en fournir les résultats aux autorités sanitaires. Un rapport détaillé des activités du CNR est ainsi fourni annuellement à Santé publique France. Il est accessible en ligne l’année suivante après validation par les autorités de tutelles.
Pour les Escherichia coli entérohémorragiques (EHEC), notamment responsables du Syndrome Hémolytique et Urémique (SHU), le CNR-ESS de l’Institut Pasteur et le CNR associé de l’hôpital Robert Debré de Paris ont contribué à la mise en place depuis 1996, d’un système de surveillance microbiologique.
Ce réseau signale en temps réel à Santé publique France :
- Tout cas confirmé d’infections à EHEC
- Un bilan ou un point de situation à tout moment par sérogroupe ou sérotype et par département.
Et adresse régulièrement à Santé publique France, en collaboration avec le CNR associé, un bilan des cas positifs d’infections à EHEC et un suivi en temps réel en cas de survenue de cas groupés ou d’épidémies.
Pour les Shigella/EIEC, et le CNR-ESS réalise une surveillance continue spatio-temporelle des cas de shigellose isolés de France métropolitaine et d’Outre-mer, basée sur les souches transmises par le réseau de laboratoires partenaires : suivi de la distribution des différents sérotypes et de la résistance aux anti-infectieux par département et par type de population (répartition par âge et sexe). Tout groupement génomique détecté est immédiatement signalé à Santé publique France.
Pour les Salmonella, le CNR-ESS exerce une surveillance continue des salmonelloses en France grâce aux souches d’origine humaine envoyées par les laboratoires (France métropolitaine et Outre-mer).
Le système informatisé et automatisé de surveillance et d’alerte des salmonelloses humaines du CNR permet de :
- documenter les tendances spatio-temporelles de tous les sérotypes sous surveillance (depuis 1992),
- détecter précocement au niveau national, régional ou départemental ou dans certaines classes d’âge (par exemple les nourrissons de moins de un an) toute augmentation anormale du nombre d’isolements de Salmonella (trois algorithmes différents utilisés),
- produire des documents (courbes, graphiques à barre,..) illustrant les tendances ou la répartition des sérotypes sous surveillance.
Il adresse à Santé publique France :
- de façon hebdomadaire, la liste des sérotypes de Salmonella à déclaration obligatoire, à savoir, les cas de salmonelloses à sérotypes majeurs que sont Typhi, Paratyphi A, B et C ainsi que les cas groupés de salmonelloses quel que soit le sérotype (épidémies familiales, hospitalières, scolaires, toxi-infections alimentaires collectives, infections collectives) qui lui sont signalés par les laboratoires correspondants,
- un relevé hebdomadaire de surveillance provisoire signalant le(s) sérotype(s) et les clusters génomiques dépassant les valeurs attendues (algorithme de détection sur la base des souches reçues au CNR),
- une notification en temps réel de tout groupement génomique détecté.
En cas de survenue d’une épidémie : le CNR-ESS communique à Santé publique France les informations épidémiologiques essentielles apportées par les laboratoires expéditeurs sur les souches impliquées.
Le délai des résultats de typage des souches traitées par WGS est de deux à trois semaines. Cependant nous vous rappelons que, quel que soit le sérotype rendu, pour un sérotype mineur, la prise en charge du patient n’est pas modifiée, car le diagnostic de salmonelle et l’antibiogramme ont été remis par vos soins au clinicien qui n’attend pas de sérotype.
La réception des souches de Salmonella et de Shigella/EIEC est essentielle pour les activités de surveillance. Nous vous encourageons à nous envoyer toute souche d’origine humaine (voir la partie « Envoyer une souche »).
COLLECTION DE SOUCHES
Toutes les souches adressées au CNR depuis 1947 ont été conservées en tubes gélosés gardés à température ambiante. La collection du CNR comprend ainsi plus de 300.000 souches. L’ensemble de tous les sérotypes connus de Salmonella est conservé sous forme lyophilisée au Centre Collaborateur OMS de référence et de Recherche sur les Salmonella (CCOMS). Les souches particulières ou de sérotype rares sont également conservées congelées à -80°C.
FORMATION
Des conseils à la fois pratiques (milieu de transport, feuille d’information...), diagnostic (importance des gènes de pathogénicité ou du sérotype détecté…), thérapeutiques et/ou épidémiologiques sont donnés par les biologistes par téléphone ou courrier électronique (pour Shigella/EIEC et Salmonella au CNR-ESS à l’Institut Pasteur : colishig@pasteur.fr ou salmonella@pasteur.fr et pour E. coli au CNR associé à l’hôpital Robert Debré : e.coli@aphp.fr).
Les biologistes participent aux réunions, missions et conférences nationales et internationales. Le CNR-ESS reçoit des stagiaires dans le cadre de projets définis, dont certains du réseau international des Instituts Pasteur.
COLLABORATIONS ET RÉSEAUX
Nationaux
- Depuis la création du CNR-ESS, plus de 2 200 laboratoires collaborateurs ont adressé des souches. Pendant le dernier mandat (2017-2021) Le nombre de laboratoires collaborateurs a diminué chaque année, surtout en lien à l’association progressive de laboratoires privés de biologie médicale en structures unies.
Année
LBM
Hospitalier Total
2017
737
497
1234
2018
594 452 1046 2019
534 447 981 2020 405 387 792 2021 317 367 684
- Avec Santé publique France, la direction générale de l’alimentation (DGAL) ou l’ANSES.
Internationales
- Avec le « European Centre for Disease Prevention and Control » (ECDC) :
Le CNR-ESS participe au Réseau Européen de surveillance de l’ECDC en répondant aux demandes d’information et en transmettant les données épidémiologiques essentielles sur les souches d’origine humaine identifiées au CNR.
- Avec l’Organisation Mondiale de la Santé (OMS) :
L’unité héberge le Centre Collaborateur de l’OMS (CCOMS) pour la Référence et la Recherche sur les Salmonella qui est en charge de la nomenclature internationale des sérotypes de Salmonella et de mettre à jour régulièrement le schéma de White-Kauffmann-Le Minor. Les cadres du CNR, également responsables du CCOMS effectuent des missions à la demande de l’OMS (workshops ou cours GFN). Le CNR-ESS transmet au CCOMS, pour homologation, tous les sérotypes nouveaux de Salmonella qu’il a identifiés.
- Avec le Réseau International des Instituts Pasteur (RIIP) :
L’Unité collabore dans des projets impliquant le RIIP en typant et sous-typant les souches isolées et en accueillant de jeunes chercheurs en formation
Par ailleurs le CNR-ESS apporte son expertise microbiologique aux laboratoires de biologie médicale et aux laboratoires de sécurité alimentaire travaillant sur les pathogènes entériques (envoi de protocoles, accueil de stagiaires qui sont formés sur les techniques de typage moléculaire).
Mis à jour le 18 avril 2024

