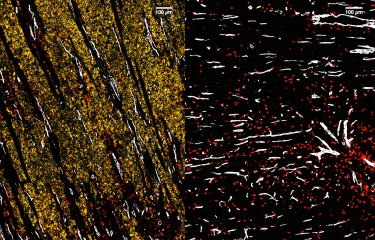
Une récente étude démontre que le pneumocoque, en modifiant durablement une protéine liée à l’ADN, impacte l’expression des gènes de certaines cellules pulmonaires. Même après élimination de la bactérie, cette modification persiste plusieurs jours et rend nos cellules plus tolérantes à de futures infections.
60 milliards de kilomètres soit 400 fois la distance Terre-Soleil : telle serait la longueur de la totalité de l’ADN du corps humain, s’il était entièrement déroulé et mis bout-à-bout. Or, les deux mètres d’ADN contenus dans chacune de nos cellules sont condensés dans un noyau de 5 à 10 micromètres de diamètre (à titre d’exemple, le diamètre d’un cheveu est de 50 micromètres environ). Comment l’ADN tient-il dans un si petit noyau ? En étant organisé sous forme de chromatine, une structure complexe composée d’acides nucléiques et de protéines. Parmi ces protéines, les histones, des molécules autour desquelles s’enroule l’ADN et qui jouent un rôle central dans le compactage de notre matériel génétique.
Les modifications persistent, même lorsque le pneumocoque est éliminé
Les histones ont également un rôle essentiel dans la régulation de l’expression des gènes : ces derniers sont moins accessibles aux molécules permettant la transcription (synthèse d’ARN à partir de l’ADN) lorsque la chromatine est plus condensée. Une modification au niveau des histones -on parle également de mécanisme épigénétique-impacte donc le niveau d’expression des gènes et par conséquent le métabolisme cellulaire.
Dans une récente étude, des chercheuses et chercheurs de l’Institut Pasteur ont étudié Streptococcus pneumoniae -ou pneumocoque- une bactérie naturellement colonisatrice des cellules de l’épithélium respiratoire. Chez les personnes fragiles, cette bactérie peut s’avérer pathogène et provoquer des pneumonies, des méningites ou des otites. Les scientifiques ont mis en évidence que le pneumocoque induit une modification chimique au niveau des histones. Cette modification impacte le niveau d’expression de plusieurs centaines de gènes et altère la physiologie des cellules. Plus surprenant encore, même en éliminant le pneumocoque via un traitement antibiotique, cette modification persiste. “Notre travail nous permet d’affirmer que la marque épigénétique est conservée au moins 9 jours après l’infection et en absence totale de bactéries. Il est même possible que cette marque perdure plusieurs semaines mais cela reste à démontrer ” explique Mélanie Hamon, responsable de l’unité Chromatine et infection.
Des cellules rendues plus permissives à une infection ultérieure
Si l’infection par le pneumocoque entraîne des modifications durables au niveau cellulaire, quelles en sont les conséquences ? Les scientifiques apportent plusieurs éléments de réponse. “Comme le tabac, le pneumocoque modifie l’épigénétique des cellules. Cette marque induit une forme de mémoire qui change la manière dont les cellules interagissent avec les bactéries” détaille Mélanie Hamon. L’autrice principale, Christine Chevalier, précise que “les cellules modifiées répondent différemment à une nouvelle infection et deviennent plus permissives à l'adhésion des bactéries.” Autrement dit, les cellules de l’épithélium pulmonaire sont fragilisées par cette première infection au pneumocoque.
Si de nombreuses questions restent en suspens, cette étude est une bonne illustration de la complexité des interactions entre les cellules de notre organisme et les microbes. Elle démontre également comment une infection, d’apparence bénigne, est capable d’influencer notre santé future. Enfin, ces travaux ouvrent également la voie à de nouvelles stratégies thérapeutiques, avec comme objectif d’empêcher la modification des histones et du taux d’expression des gènes de nos cellules.
Source : Epithelial cells maintain memory of prior infection with Streptococcus pneumoniae through di-methylation of histone H3, Nature communications, 2 juillet 2024
Christine Chevalier1, Claudia Chica2, Justine Matheau1,3, Adrien Pain2, Michael G. Connor1 & Melanie A. Hamon1
1Institut Pasteur, Université Paris Cité, Chromatin and Infection Laboratory, F-75015 Paris, France.
2 Institut Pasteur, Université Paris Cité, Bioinformatics and Biostatistics Hub, F-75015 Paris, France.
3 Bio Sorbonne Paris Cité doctoral school, Department of Infectiology Microbiology, Université Paris Cité, F-75006 Paris, France.