La résistance des bactéries aux antibiotiques est un phénomène qui s’accentue, créant un problème de santé publique majeur. Parmi les possibles alternatives aux antibiotiques, l’utilisation de virus infectant les bactéries, les phages. Une récente étude démontre néanmoins que l’un des principaux mécanismes de résistance utilisés par les bactéries contre les antibiotiques est également utilisé contre les phages. Ce travail souligne l’importance d’élaborer des stratégies complémentaires contre l’antibiorésistance et ouvre la voie au développement d’outils biotechnologiques.
Apparues sur Terre il y a 3,5 à 4 milliards d’années, les bactéries se sont rapidement dotées de mécanismes d’adaptation leur permettant de survivre dans les environnements changeants et parfois hostiles qui ont émaillé l’histoire de notre planète. Modification ponctuelle et aléatoire de leur matériel génétique (mutations), faculté à capturer des gènes de l’environnement ou issus d’autres micro-organismes (transfert horizontal de gènes), capacité à réarranger leur génome en déplaçant au sein de celui-ci des séquences d’ADN mobiles, etc : les bactéries peuvent mettre en œuvre de nombreux processus pour s’adapter à leur environnement.
Les intégrons : un outil adaptatif pour résister aux antibiotiques
C’est d'ailleurs cette capacité d'adaptation qui permet aujourd’hui à de plus de plus de bactéries de devenir résistantes aux antibiotiques. Utilisés en médecine courante depuis le milieu du XXe siècle contre les infections bactériennes, les antibiotiques s’avèrent de moins en moins efficaces. Demain, une maladie bénigne pourrait devenir beaucoup plus difficile à traiter. Si rien n’est fait, en 2050, les infections bactériennes résistantes aux antibiotiques pourraient provoquer la mort de 10 millions de personnes par an dans le monde selon l’OMS.
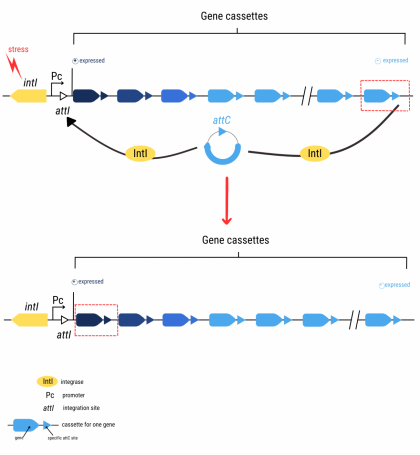
“Chez certains groupes de bactéries, les intégrons sont les principaux vecteurs de résistance aux antibiotiques" explique Didier Mazel, responsable de l’unité Plasticité du génome bactérien à l’Institut Pasteur. Les intégrons sont en quelque sorte "une boîte à outils génétique” précise Céline Loot, chercheuse CNRS dans la même unité de recherche. Cette boîte à outils se compose :
- d'un gène codant pour une intégrase (IntI), une enzyme clivant l’ADN à des endroits spécifiques
- d’un promoteur (Pc), une petite séquence d’ADN située en amont d’un gène et indispensable à son expression
- de cassettes de gènes, séquences d’ADN mobiles, contenant généralement un gène, et terminées par des sites spécifiques attC
- d’un site d’intégration appelé attI
Dans certaines conditions de stress, par exemple en cas de présence d’antibiotiques, la bactérie va exprimer l’intégrase. Cette protéine va alors cliver la cassette de gène au niveau des sites attC permettant son excision. Cette cassette excisée va ensuite s’intégrer au niveau du site attI, près du promoteur Pc, permettant alors l’expression du gène porté par ladite cassette. Le génome bactérien est alors réarrangé et la bactérie acquiert la caractéristique liée au gène dorénavant exprimé.
Si la présence de gènes de résistance aux antibiotiques au sein des cassettes est connue, nombre d’entre elles portent des gènes aux fonctions inconnues. Des scientifiques de l’Institut Pasteur, du CNRS, de l’Université Paris Cité et de l’Université de Montréal ont voulu en savoir plus. Résultat ? Ils ont découvert qu’un pourcentage non-négligeable de ces cassettes contiennent des systèmes de défense contre les phages.
Phages, antibiotiques : même combat !
Ici, précisions et petit retour en arrière. Les phages sont des virus infectant uniquement les bactéries. Piste prometteuse contre les infections bactériennes au début du XXe siècle, ils ont été supplantés par l’adoption massive des antibiotiques au tournant des années 1940-1950, au point que les recherches sur ces virus sont plus ou moins tombées en désuétude. Mais, l’antibiorésistance croissante marque depuis plusieurs années un regain d’intérêt pour ces virus qui pourraient être des substituts thérapeutiques contre les bactéries pathogènes.
Pour la première fois donc, des chercheurs et chercheuses démontrent que les intégrons sont également impliqués dans la résistance aux phages. En d’autres termes, les bactéries utilisent les mêmes armes adaptatives contre les antibiotiques et les virus qui les infectent ! L’ampleur du phénomène va même au-delà de ce qu’envisageaient les auteurs de l’étude. “Nous estimons qu’au moins 10% des cassettes d’intégron codent pour des systèmes anti-phage. Nous n’imaginions pas un pourcentage aussi élevé. C’est précisément pour cela que nous parlons de biobanque.” relate Eduardo Rocha chercheur CNRS et responsable de l’unité Génomique évolutive des microbes à l’Institut Pasteur. Il y aurait ainsi plusieurs milliers de cassettes anti-phage, faisant des intégrons un immense réservoir de gènes contre ces virus.
Les scientifiques ont par ailleurs cherché à caractériser ces systèmes anti-phage. Ils ont ainsi mis en évidence seize systèmes anti-phage fonctionnels chez Vibrio cholerae et E. coli, et jusqu’alors inconnus. Ces systèmes sont relativement petits, sans doute contraints par le plus faible taux de recombinaison des cassettes de grande taille. Les chercheurs et chercheuses ont même découvert dans un de ces systèmes, la plus petite protéine conférant une résistance aux phages connue à ce jour. Par ailleurs, en se basant sur les modèles prédictifs de structure des protéines impliquées dans ces systèmes, les scientifiques ont trouvé des domaines communs à d’autres molécules de défense. Des protéines dont le rôle est d’arrêter la croissance bactérienne ou d’engendrer la mort cellulaire afin d’empêcher la multiplication des phages. Ces fonctionnalités ont ensuite été démontrées expérimentalement pour les systèmes anti-phage nouvellement décrits.
Demain, quelles thérapies contre les bactéries pathogènes ?
Ces résultats ouvrent la voie à de nouveaux outils biotechnologiques. Cette étude pourrait par exemple contribuer à mieux protéger des infections phagiques les bactéries utilisées dans les processus industriels. Elle permet également de concevoir de nouvelles pistes de recherche. “Nous étudions actuellement au laboratoire la dynamique de résistance aux phages médiée par les intégrons. Les résultats sont encore très préliminaires. Il n’est pas impossible que la différence de nature entre les antibiotiques et les phages se traduisent par des dynamiques de résistance bactérienne différentes” explique Céline Loot. Plus globalement, selon la chercheuse, cette étude “ne remet pas en question l’utilisation des phages contre les bactéries pathogènes” car “les problèmes d’évolution de la résistance sont inévitables pour toute approche thérapeutique.”
Un processus d’évolution inévitable, qui suggère que la meilleure stratégie contre la résistance bactérienne est sans doute plurielle. “Il est effectivement probable qu’une approche combinée soit la plus efficace” abonde Frédérique Le Roux, chercheuse à l’Université de Montréal. "Contre des bactéries pathogènes, nous pouvons, par exemple, imaginer un traitement contenant des antibiotiques associés à des cocktails de phages. L’approche CRISPR pourrait aussi être utilisée. Il y a quelques années, nous avons également mis au point une stratégie basée sur l’expression de toxines dans les bactéries pathogènes, en détournant leurs capacités à s’échanger des gènes via des plasmides, des séquences d’ADN relativement petites. Il existe des alternatives prometteuses. ” précise Didier Mazel.
Bref, même si ces travaux sont une nouvelle illustration des formidables capacités adaptatives des bactéries, dans la lutte contre leur résistance, les scientifiques n’ont pas dit leur dernier mot !
Sedentary chromosomal integrons as biobanks of bacterial anti-phage defense systems, Science, 8 mai 2025
Baptiste Darracq1,2†, Eloi Littner2,3,4†, Manon Brunie1,2, Julia Bos1, Pierre Alexandre Kaminski1, Florence Depardieu5, Weronika Slesak1, Kevin Debatisse1, Marie Touchon3, Aude Bernheim6, David Bikard5, Frédérique Le Roux7*, Didier Mazel1*, Eduardo P.C. Rocha3‡* and Céline Loot1‡*
1Institut Pasteur, Université Paris Cité, Unité Plasticité du Génome Bactérien, CNRS, UMR3525, 75724 Paris, France.
2Sorbonne Université, Collège doctoral, F-75005, Paris, France.
3Institut Pasteur, Université Paris Cité, Microbial Evolutionary Genomics, CNRS UMR3525, 75724 Paris, France.
4DGA CBRN Defence, 91710 Vert-le-Petit, France.
5Institut Pasteur, Université Paris Cité, Synthetic Biology, CNRS UMR3525, 75724 Paris, France.
6Institut Pasteur, Université Paris Cité, Molecular Diversity of Microbes lab, CNRS UMR3525, 75724 Paris, France.
7University of Montréal, Faculty of medicine, Department of microbiology, infectiology and immunology, Montreal, QC H3T 1J4, Canada.
†These authors contributed equally to this work
‡ These authors contributed equally to this work
*Corresponding authors