Le séquençage du génome de souches du virus Ebola circulant en Guinée par des chercheurs de l’Institut Pasteur de Dakar et de Paris, du CNRS et de l’université de Sydney a permis de retracer la diffusion du virus et de suivre son évolution dans le pays où l’épidémie a débuté. Ces travaux révèlent la co-circulation, notamment dans les régions urbaines de la capitale du pays et des villes voisines, de trois différents variants du virus dont les mutations sont décrites dans un article paru dans Nature. La caractérisation des variations génétiques du virus est cruciale pour s’assurer de l’efficacité continue des outils de diagnostic, et pour le développement de traitements et de vaccins efficaces.
L’épidémie d’Ebola sévit en Afrique de l’Ouest depuis plus d’un an : 27 341 cas et 11 184[1] ont été rapportés. Le point de départ de l’épidémie a été identifié dans une région forestière de Guinée (au Sud-Est du pays), mais très rapidement la capitale guinéenne a été touchée. Dès mars 2014, l’Institut Pasteur de Dakar a installé un laboratoire mobile au sein de l’hôpital de Donka (Conakry) pour contribuer aux efforts de diagnostic en Guinée. Cette implication de volontaires de l’Institut Pasteur et de son réseau, aussi mobilisés dans d’autres régions de Guinée comme à Macenta, s’est poursuivie durant toute la durée de l’épidémie.
Pour mettre au point de meilleures stratégies de traitement, concevoir des vaccins efficaces, et s’assurer de l’efficacité continue des outils de diagnostic, il est essentiel de suivre l’évolution du génome du virus Ebola. Dans cette optique, des scientifiques de l’Institut Pasteur de Dakar et de Paris, du CNRS et de l’Université de Sydney ont contribué aux efforts internationaux en caractérisant les isolats du virus Ebola ayant circulé en Guinée entre juillet et novembre 2014.
Une collaboration établie avec des chercheurs du Broad Institue (Cambridge, Etats-Unis), déjà mobilisés en Sierra Leone, a permis d’optimiser le séquençage du virus contenu dans des échantillons rares et contenant très peu de matériel biologique. L’analyse de ces séquences révèle l’existence de trois variants distincts du virus qui ont circulé en même temps en Guinée, une dynamique très différente de ce qui avait été décrit pour la Sierra Leone et le Libéria.
Le premier variant (GUI-1) (en rouge sur la carte) est très proche des premiers échantillons datant du début de l’épidémie (mars 2014). Ce variant se retrouve uniquement en Guinée, à la fois dans les régions urbaines (Conakry) et les régions forestières.
Le deuxième variant (GUI-2) (en bleu sur la carte) présente un lien de parenté avec des virus circulant en Sierra Leone mais pourrait correspondre à des virus ayant évolué parallèlement en Guinée. Ces séquences représentent le chaînon manquant ayant mené aux deux introductions d’Ebola au Mali, en octobre et novembre 2014.
Le troisième variant (SLE-GUI-3) (en orange sur la carte) a été détecté à Conakry et dans des villes voisines (Forecariah, Dalaba, Coyah). Ses très grandes similarités avec des virus trouvés en Sierra Leone s’ajoutent aux données épidémiologiques pour mettre en lumière de multiples réintroductions du virus Ebola depuis la Sierra Leone vers la capitale Guinéenne.
Chaque variant est défini par une combinaison de mutations affectant différentes protéines du virus : notamment la protéine VP35 qui pourrait être un facteur de virulence, la glycoprotéine d’enveloppe du virus, ce qui pourrait moduler la perception du virus par le système immunitaire, ou encore la polymérase, qui est pourtant une région généralement plus conservée chez les virus.
Si cette étude met en lumière la diversité génétique des virus circulant au cours de l’épidémie en Guinée, la vitesse d’acquisition des mutations est tout à fait dans les marges déjà décrites pour ce genre de virus. Le suivi des variations du virus complète aussi les études épidémiologiques qui retracent les chaînes de transmission, et permettra de mieux adapter les stratégies de réponse face à de potentielles nouvelles épidémies. Enfin, l’étude des différentes mutations, déjà mises à disposition de la communauté scientifique, va permettre d’optimiser les traitements et les vaccins en cours de développement.
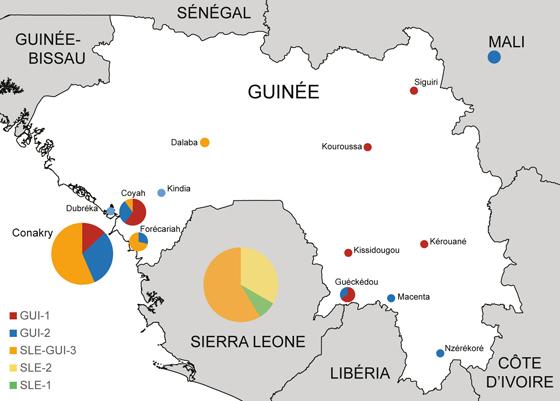
Représentation des variants du virus Ebola circulant en Guinée et Sierra Leone. Creative Commons - Nature
Evolution dans le temps du nombre de cas d'Ebola.
Source
Distinct lineages of Ebola virus in Guinea during the 2014 West African epidemic, Nature, DOI : 10.1038/nature14612, 24 juin 2015
Etienne Simon-Loriere1,2†*, Ousmane Faye3†, Oumar Faye3†, Lamine Koivogui4, Nfaly Magassouba5, Sakoba Keita6, Jean-Michel Thiberge7, Laure Diancourt7, Christiane Bouchier8, Matthias Vandenbogaert7, Valérie Caro7, Gamou Fall3, Jan P. Buchmann9, Christan B. Matranga10, Pardis C. Sabeti10,11, Jean-Claude Manuguerra7, Edward C. Holmes9‡* and Amadou A. Sall3‡*.
1 Institut Pasteur, Functional Genetics of Infectious Diseases Unit, 75724 Paris Cedex 15, France.
2 CNRS URA3012 Hôtes, vecteurs et agents infectieux : biologie et dynamique, Paris 75015, France.
3 Institut Pasteur de Dakar, Arbovirus and Viral Hemorrhagic Fever Unit, Dakar, Senegal.
4 Institut National de Santé Publique de Guinée, Conakry, Guinée.
5 Projet de fièvres hémorragiques de Guinée, Université Gamal Abdel Nasser, Conakry, Guinée.
6 Ministry of Health, Conakry, Guinea.
7 Institut Pasteur, Unité Environnement et Risques Infectieux, Cellule d’Intervention Biologique d’Urgence, 75724 Paris Cedex 15, France.
8 Institut Pasteur, Genomic platform, 75724 Paris Cedex 15, France.
9 Marie Bashir Institute for Infectious Diseases and Biosecurity, Charles Perkins Centre, School of Biological Sciences and Sydney Medical School, The University of Sydney, Sydney, NSW 2006 Australia.
10 Broad Institute, 75 Ames Street, Cambridge 02142, MA, USA.
11 FAS Center for Systems Biology, Department of Organismic and Evolutionary Biology, Harvard University, 52 Oxford Street, Cambridge 02138, MA, USA.
† Ces auteurs ont contribué de manière équivalente au travail.
‡ Ces auteurs ont conjointement supervisé ce travail.