En collaboration avec New England Biolabs, le groupe de recherche Réplication de l’ADN dirigé par Ludovic Sauguet à l’Institut Pasteur, a publié dans Nucleic Acids Research une approche innovante basée sur le séquençage longue lecture PacBio pour caractériser avec une précision inédite les erreurs des ADN polymérases.
Les ADN polymérases (DNAPs), enzymes essentielles à la réplication et à la réparation de l'ADN, constituent le socle de nombreuses applications biotechnologiques, de la PCR au séquençage en passant par le clonage et les thérapies géniques. Malgré leur remarquable précision, ces enzymes commettent occasionnellement des erreurs aux conséquences potentiellement importantes, qu'elles soient évolutives ou pathologiques, notamment dans le développement de cancers. Comprendre les mécanismes sous-jacents à ces erreurs représente donc un enjeu majeur pour améliorer nos connaissances mais aussi optimiser leur utilisation.
Perspectives biotechnologiques prometteuses
Ces travaux ouvrent des perspectives intéressantes pour la conception d’enzymes sur mesure, adaptées à des applications spécifiques nécessitant soit une fidélité accrue, soit au contraire une mutagénèse contrôlée, avec des applications dans les domaines biotechnologiques, thérapeutiques et diagnostiques.
Une approche méthodologique inédite
L'innovation majeure de cette étude réside dans l'utilisation d'une méthode à haut débit exploitant le séquençage longue lecture Pacific Biosciences (PacBio), sans recours à l'amplification PCR. Cette approche, non biaisée par la séquence de l’ADN matrice au contraire des autres techniques décrites jusqu’ici, permet de mesurer directement les taux d'erreur et de cartographier finement les profils d'erreurs spécifiques à chaque enzyme. Les DNAPs réplicatives possèdent en effet une double activité catalytique : une activité polymérase pour synthétiser l'ADN et une activité exonucléase de relecture pour éliminer les nucléotides mal incorporés. La méthode développée permet d'évaluer l'efficacité de ces deux mécanismes de manière intégrée.
Des profils d'erreurs famille-spécifiques révélés
Les chercheurs ont analysé les quatre principales familles d'ADN polymérases réplicatives (A, B, C et D), révélant des profils d'erreurs diversifiés et spécifiques à chaque famille. Cette caractérisation fine met en évidence des mécanismes moléculaires distincts gouvernant la fidélité de ces enzymes, offrant ainsi une compréhension approfondie de leurs modes de fonctionnement respectifs.
Facteurs influençant la fidélité enzymatique
L'étude a également identifié plusieurs paramètres critiques qui affectent la précision des DNAPs. Par exemple, les ratios des 4 désoxyribonucléotides triphosphates qui rentrent dans la composition de l’ADN (dATP, dGTP, dCTP, dTTP) dans le milieu réactionnel influencent significativement le taux d'erreur, suggérant que l'équilibre des substrats disponibles module directement la fidélité de réplication. Les composants du système de réplication et les mutations dans les sites actifs de l'enzyme - tant au niveau du domaine polymérase que de l'exonucléase - ont également été explorés, révélant comment des modifications structurelles peuvent altérer ou améliorer la précision enzymatique.
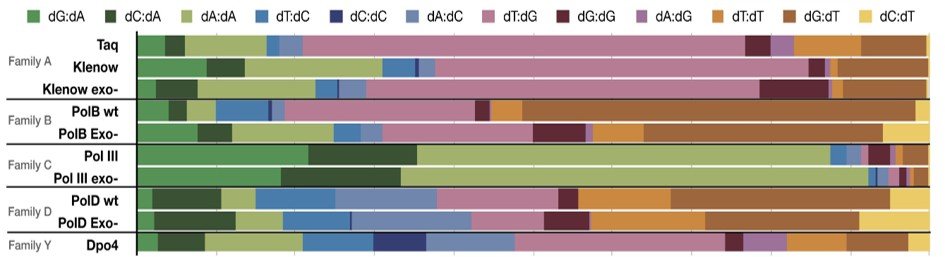
DNA polymerase error rates and profiles. (A) Fidelity measurements in substitutions in a million bases for the different families of DNA polymerases and their respective exo- mutant. (B) Substitution spectra for each DNA polymerase, with dA misincorporations in green, dC misincorporations in blue, dG misincorporations in pink, and dT misincorporations in orange. Legend corresponds to template strand:nascent strand mispair (i.e. dG:dA = template strand dG, nascent strand dA).





