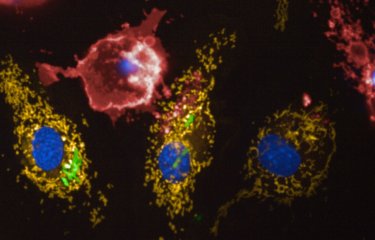
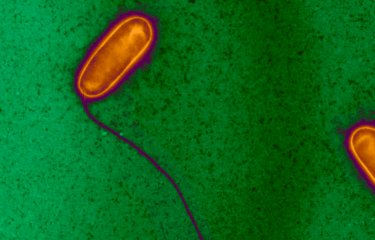
Des chercheurs de l'Institut Pasteur et du CNRS, en collaboration avec le Centre National de Référence des Légionelles (Inserm) à Lyon, ont comparé le contenu génomique de dizaines de souches de la bactérie responsable de la « maladie du légionnaire ». Leur étude, publiée dans Genome Research, ouvre la voie à la mise au point de tests de diagnostic rapide, qui font aujourd'hui défaut pour la surveillance de l'environnement et donc pour une prévention efficace de la légionellose.
Communiqué de presse
Paris, le 6 février 2008
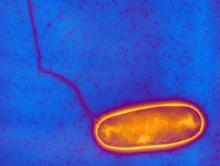
Une étude menée par Carmen Buchrieser, chef de l’unité de Biologie des bactéries intracellulaires (CNRS URA 2171) de l’Institut Pasteur, en collaboration avec le Centre National de Référence des Légionelles (Inserm U851), visait à comparer les génomes de différentes souches de Legionella.
L’analyse génomique a porté sur 217 souches de Legionella pneumophila, l’espèce pathogène pour l’homme, et 32 souches de Legionella non pneumophila, isolées des patients et de l’environnement.
L’étude a notamment permis d’identifier trois gènes spécifiques de Legionella pneumophila sérogroupe 1, groupe responsable à lui seul de 84% des cas de légionellose dans le monde.
Elle a aussi montré que la souche Paris, qui avait provoqué une épidémie nosocomiale dans un hôpital parisien en 2000, était une souche responsable de nombreux cas sporadiques et épidémies dans le monde. Là encore, des gènes spécifiques ont été identifiés.
« La connaissance de ces gènes va désormais nous permettre de développer des techniques de diagnostic plus fines, plus rapides et plus performantes », souligne Carmen Buchrieser.
Sur la base de leurs résultats, les chercheurs travaillent en effet à développer un test de PCR (1) en temps réel qui permettra une détection spécifique et sensible de Legionella pneumophila, et parallèlement un typage rapide et fiable du sérogroupe 1 de cette bactérie dans des échantillons d’eau des hôpitaux et de l’environnement. Un test qui permettrait d’identifier une telle souche en quelques heures alors que plusieurs jours sont aujourd’hui nécessaires.
____________________________
(1) Polymerase Chain Reaction : technique d’amplification génique in vitro permettant d’obtenir d’importantes quantités d’un fragment d’ADN spécifique et de longueur définie.
Source
"Multi-genome analysis identifies a worldwide distributed epidemic Legionella pneumophila clone that emerged within a highly diverse species »: Genome Research, 6 février 2008
C. Cazalet (1,2), S. Jarraud (3), Y. Ghavi-Helm (1,2), F. Kunst (1,2), P. Glaser (1,2), J. Etienne (3) et C. Buchrieser (1,2,4)
1. Unité de Génomique des microorganismes pathogènes, Institut Pasteur, Paris
2. CNRS URA 2171, Paris
3. Centre National de Référence des Légionelles, Laboratoire de bactériologie, Inserm U851, Faculté de médecine, Lyon
4. Unité postulante des Bactéries intracellulaires, Institut Pasteur, Paris
Contacts presse
Service de presse Institut Pasteur
Corinne Jamma ou Nadine Peyrolo
01 40 61 33 41 – cjamma@pasteur.fr
Service de presse CNRS
Laetitia Louis - 01 44 96 51 37 – laetitia.louis@cnrs-dir.fr
Service de presse Inserm
Séverine Ciancia – 01 44 23 60 86 – presse@tolbiac.inserm.fr