L’ADN est composé de bases azotées représentées par les lettres A, T, G, C. Celles-ci sont la base du code génétique, et présentes chez tous les êtres vivants. Cependant, chez un bactériophage, une autre base, représentée par la lettre Z, existe. Cette exception, la seule observée jusqu’à maintenant, est longtemps restée un mystère. Des chercheurs de l’Institut Pasteur et du CNRS, en collaboration avec le CEA, ont désormais élucidé la voie de biosynthèse de cette base. Ces travaux ont été publiés dans Science le 30 avril 2021.
L’ADN, ou acide désoxyribonucléique, est le support de l’information génétique chez tous les organismes vivants. Cette molécule est une double hélice caractérisée par l’alternance de bases azotées purine (adénine, guanine) ou pyrimidine (cytosine, thymine). Les bases de chaque brin d’ADN sont localisées vers le centre de l’hélice et celles-ci se lient entre elles, rassemblant ainsi les deux brins d’ADN. L’adénine forme deux liaisons hydrogène avec la thymine (A:T), et la guanine, trois liaisons hydrogène avec la cytosine (G:C). Cela vaut pour tous les êtres vivants, à une exception.
Le cyanophage S-2L, une exception aux modèles classique de la génétique
Le cyanophage S-2L est un bactériophage, c’est-à-dire un virus qui infecte les bactéries. Chez ce phage, l’adénine est complétement remplacée par une autre base, la 2-aminoadénine (représentée par la lettre Z). Celle-ci forme trois liaisons hydrogènes avec la thymine (Z :T), au lieu des deux entre l’adénine et la thymine. Ce nombre de liaisons supérieur augmente la stabilité de l’ADN à haute température, et modifie sa conformation. L’ADN est donc moins reconnu par les protéines et petites molécules.
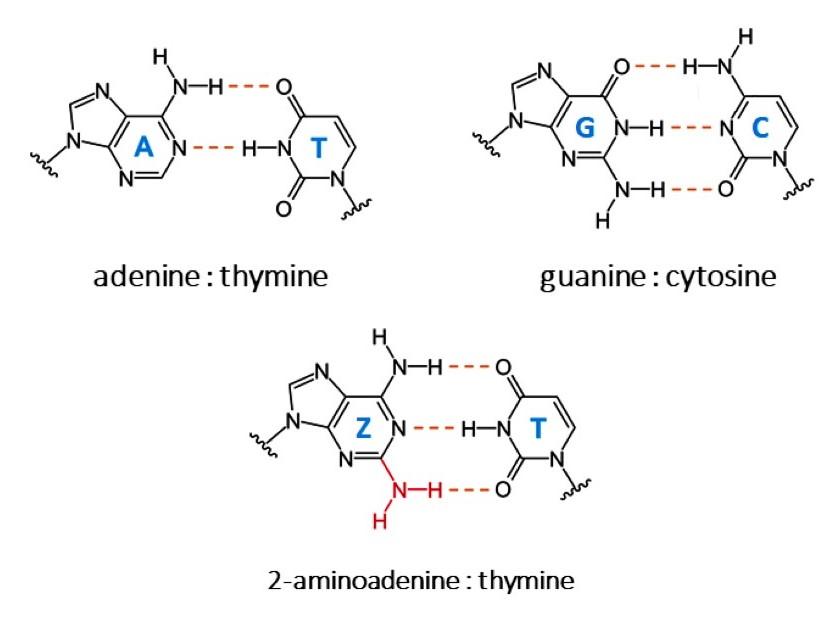
Les liaisons A : T, G : C et Z : T
La voie de biosynthèse de la 2-aminoadenine élucidée
Depuis sa découverte en 1977, le cyanophage S-2L était resté l’unique exception connue et la voie de biosynthèse de la 2-aminoadénine inconnue. Des chercheurs de l’Institut Pasteur et du CNRS, en collaboration avec le CEA, ont élucidé cette voie de biosynthèse et montré que celle-ci avait une origine enzymatique. Cela s’est fait grâce à l’identification d’un homologue d’une enzyme connue appelée adénylosuccinate synthétase, ou PurA, dans le génome du cyanophage S-2L. Une analyse phylogénétique de cette famille d’enzymes a montré un lien entre cet homologue, appelé PurZ, et l’enzyme PurA des archées. Cela indique que l’apparition de cet homologue est ancienne, et que celui-ci apportait probablement un avantage évolutif. Ces travaux ont été réalisés grâce à la plateforme de cristallographie de l’Institut Pasteur.
Cette nouvelle paire de bases Z:T et la découverte de la voie de biosynthèse de la base Z montrent qu’on peut implanter de façon enzymatique de nouvelles bases dans le matériel génétique. Cela élargit le nombre de bases codantes dans l’ADN et ouvre donc la voie au développement de biopolymères génétiques synthétiques.
Source
A third purine biosynthetic pathway encoded by aminoadenine-based viral DNA genomes, Science, 30 avril 2021
Dona Sleiman1†, Pierre Simon Garcia2,3†, Marion Lagune1, Jerome Loc’h4, Ahmed Haouz5, Najwa Taib2,6, Pascal Röthlisberger7, Simonetta Gribaldo2,Philippe Marlière8, Pierre Alexandre Kaminski1*
1Biology of Gram-Positive Pathogens, Institut Pasteur, CNRS- UMR 2001, Paris, France.
2Evolutionary Biology of the Microbial Cell, Institut Pasteur, CNRS-UMR 2001, Paris, France.
3Stress Adaptation and Metabolism, Institut Pasteur, CNRS-UMR 2001, Paris, France.
4Structural Dynamics of Macromolecules, Institut Pasteur, CNRS-UMR 3528, Paris, France.
5Crystallography Platform, C2RT, Institut Pasteur, CNRS-UMR 3528, Paris, France.
6Hub Bioinformatics and Biostatistics, Institut Pasteur, Paris, France.
7Bioorganic Chemistry of Nucleic Acids, Institut Pasteur, CNRS-UMR 3523, Paris, France.
8Génomique Métabolique, Genoscope, Institut François Jacob, CEA, CNRS, Université Evry, Université Paris-Saclay, Evry, France.
†These authors contributed equally to this work.
*Corresponding author.